エピジェネティック制御を
組織レベルで俯瞰する
エピジェネティック制御を
組織レベルで俯瞰する
- Research
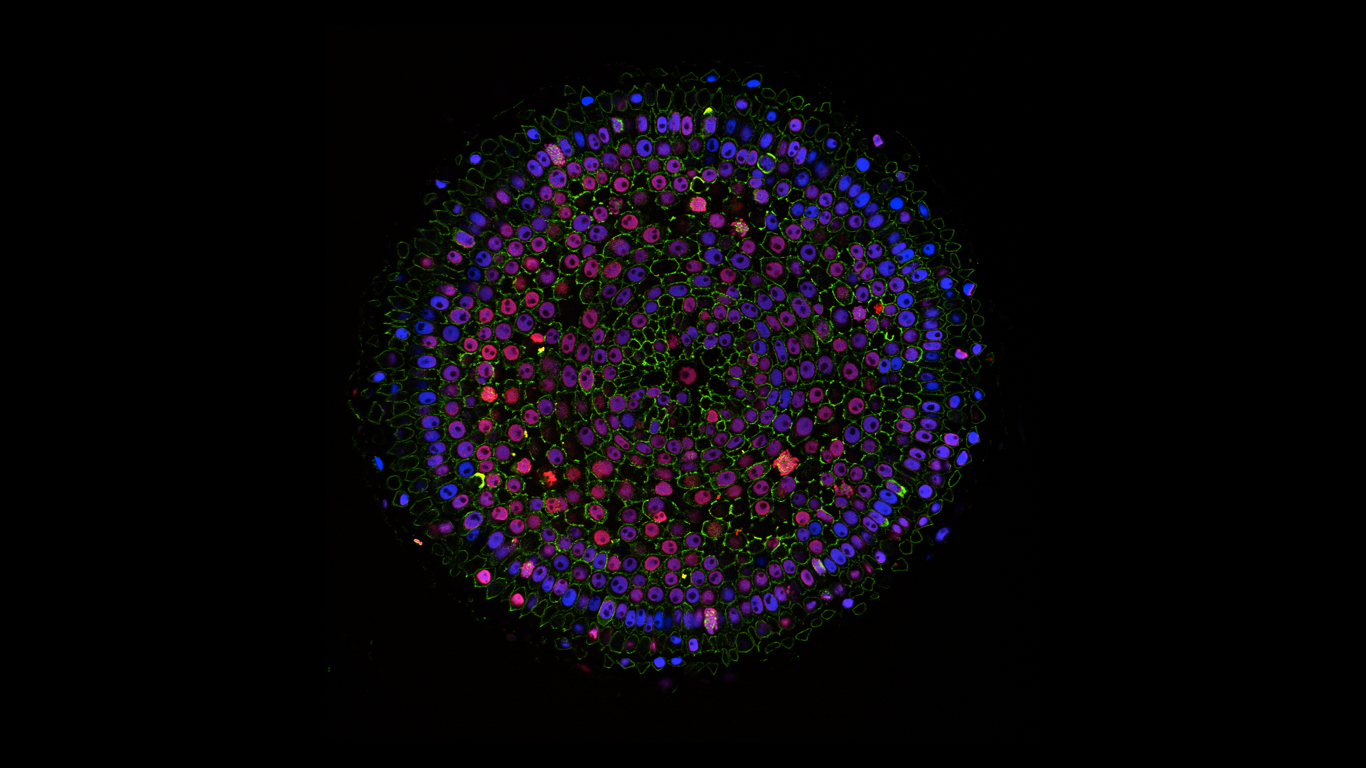
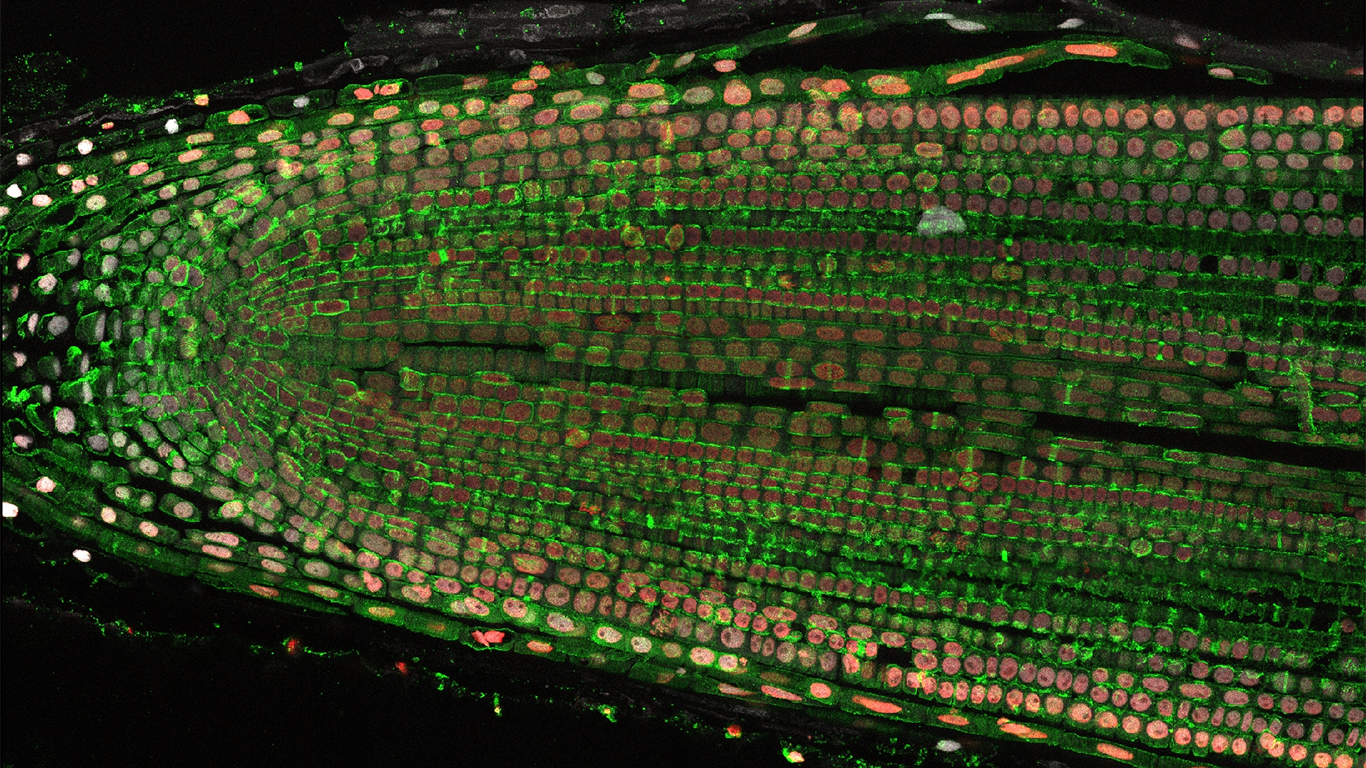
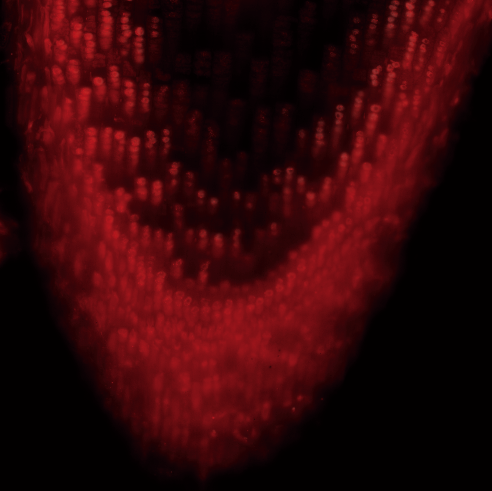
植物組織内でエピジェネティック修飾を可視化する
組織切片や透明化した組織内でエピジェネティック修飾を検出可能な組織免疫染色系を開発してきました。モデル植物に限らず、作物を含む多くの植物種に利用可能です。
参考論文
Nagaki K., Yamaji N., Murata M. (2017) ePro-ClearSee: a simple immunohistochemical method that does not require sectioning of plant samples. Scientific reports 6: srep42203.
Nagaki, K., Tanaka, K., Yamaji, N., Kobayashi, H. and Murata, M. (2015) Sunflower centromeres consist of a centromere-specific LINE and a chromosome-specific tandem repeat. Front Plant Sci, 6, 912.
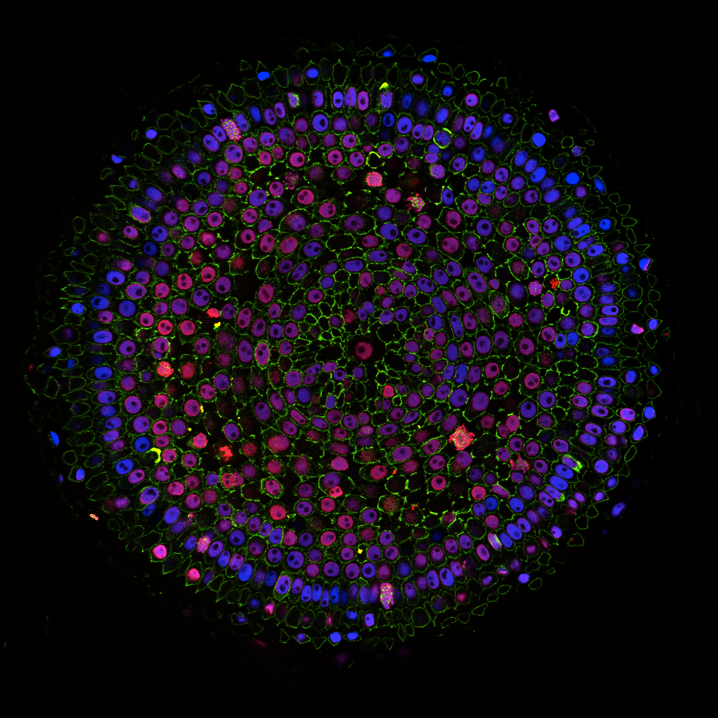
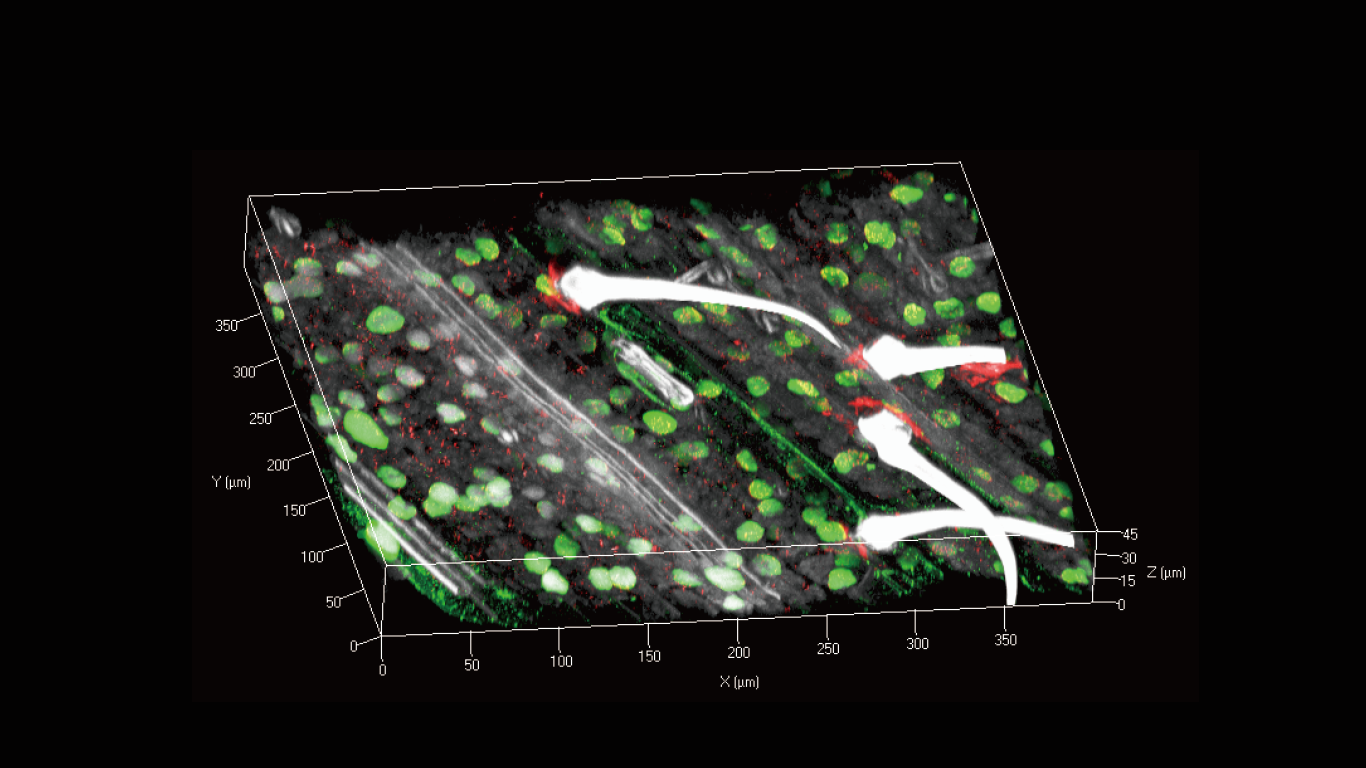
植物組織内で標的DNA配列を可視化する
組織切片や透明化した組織内でDNA配列を検出可能な方法を開発しています。従来から標的DNA配列の可視化に用いられてきた蛍光in situハイブリダイザーション(FISH)法は、DNAの1本鎖化のための熱変性が必要とされ、この操作が組織構造に影響を与えるため使用できません。そのため、低ダメージなDNA配列検出方が必要とされています。この方法と上記のエピジェネティック修飾可視化法を組み合わせることにより、組織内の個々の細胞内の目的配列のもつエピジェネティック修飾を解析することが可能となります。
参考論文
なし
ChIP & more
クロマチン免疫沈降(ChIP)法等、これまでエピジェネティック修飾解析に使われてきた手法も高解像度解析として必要です。ChIPに関する共同研究のご提案もお待ちしております。
参考論文
Nagaki, K., Tanaka, K., Yamaji, N., Kobayashi, H. and Murata, M. (2015) Sunflower centromeres consist of a centromere-specific LINE and a chromosome-specific tandem repeat. Front Plant Sci, 6, 912.
Nagaki, K., Cheng, Z., Ouyang, S., Talbert, P.B., Kim, M., Jones, K.M., Henikoff, S., Buell, C.R. and Jiang, J. (2004) Sequencing of a rice centromere uncovers active genes. Nat Genet, 36, 138-145.

- Member
班代表
准教授:長岐 清孝 Assoc. Prof. NAGAKI, Kiyotaka
E-mail:nagaki@(@以下はokayama-u.ac.jp を付けてください)
所内班員
准教授:山地 直樹 Assist. Prof. YAMAJI, Naoki
准教授:池田 陽子 Assist. Prof. IKEDA, Yoko
学外共同研究者
東京大学特任准教授:朽名 夏麿 Assist. Prof. KUTSUNA, Natsumaro
- Publications
Habu, Y., Ando, T., Ito, S., Nagaki, K., Kishimoto, N., Taguchi-Shiobara, F., Numa, H., Yamaguchi, K., Shigenobu, S., Murata, M., Meshi, T., and Yano, M. 2015. Epigenomic modification in rice controls meiotic recombination and segregation distortion. Molecular Breeding 35: 644-653
Ikeda, Y. and Nishimura, T. CHAPTER 2: The role of DNA methylation in transposable element silencing and genomic imprinting. 2015. Nuclear Functions in Plant Transcription, Signaling and Development. pp13-29, Springer.
2014年以前の論文については、コチラをご覧ください。
長岐清孝准教授の全論文リスト(Elsevier Pureへのリンク)
山地直樹准教授の全論文リスト(Elsevier Pureへのリンク)
池田陽子准教授の全論文リスト(Elsevier Pureへのリンク)