Object detector
Overview
Object detectorの概要
Object detectorでは、画像中の個々の物体を検出することができます。
例えば、画像中の個々の物体を分裂細胞“Chromosomes”とその他“others”として検出します。
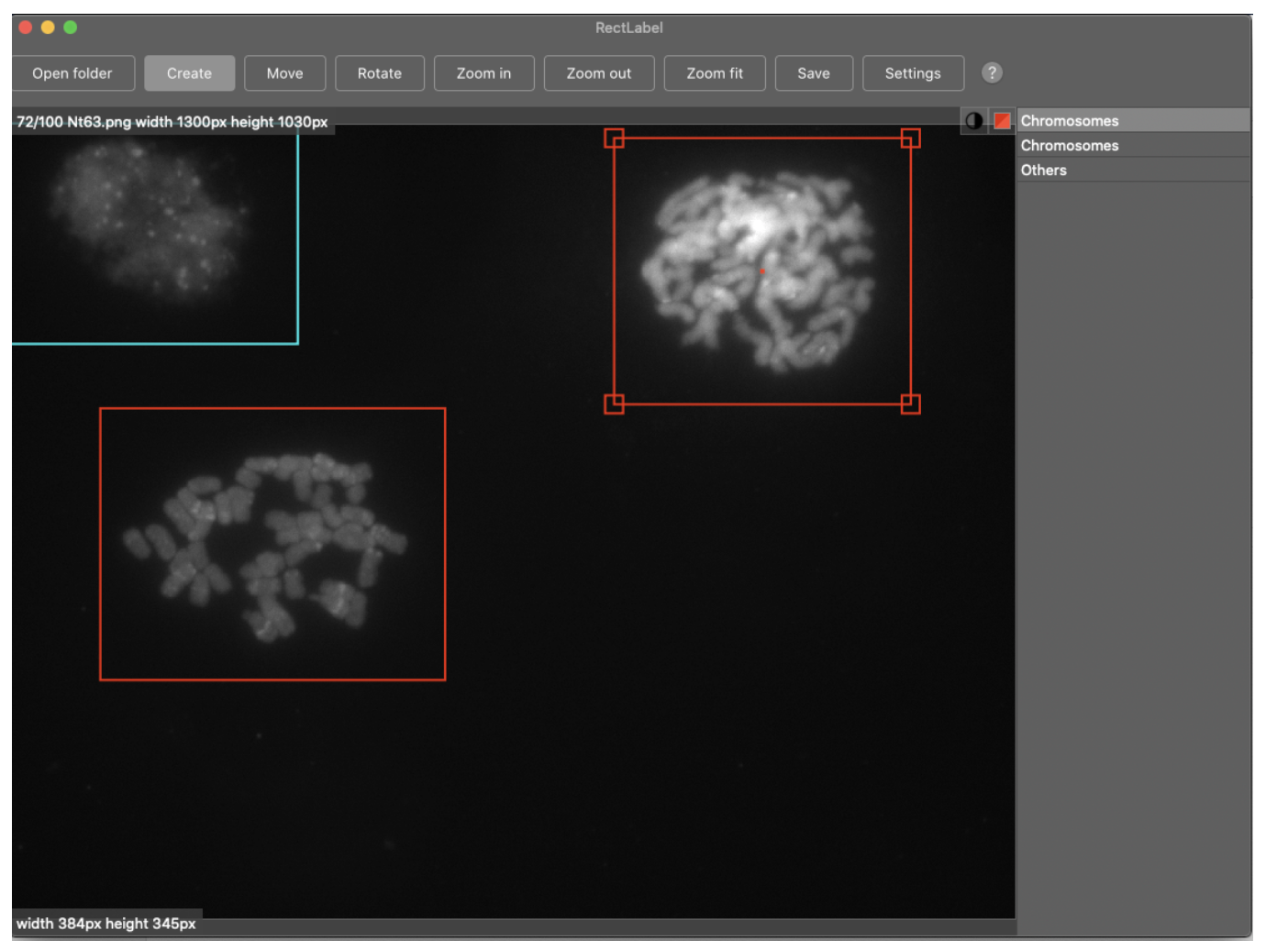
Annotations for Learning
学習のためのアノテーション
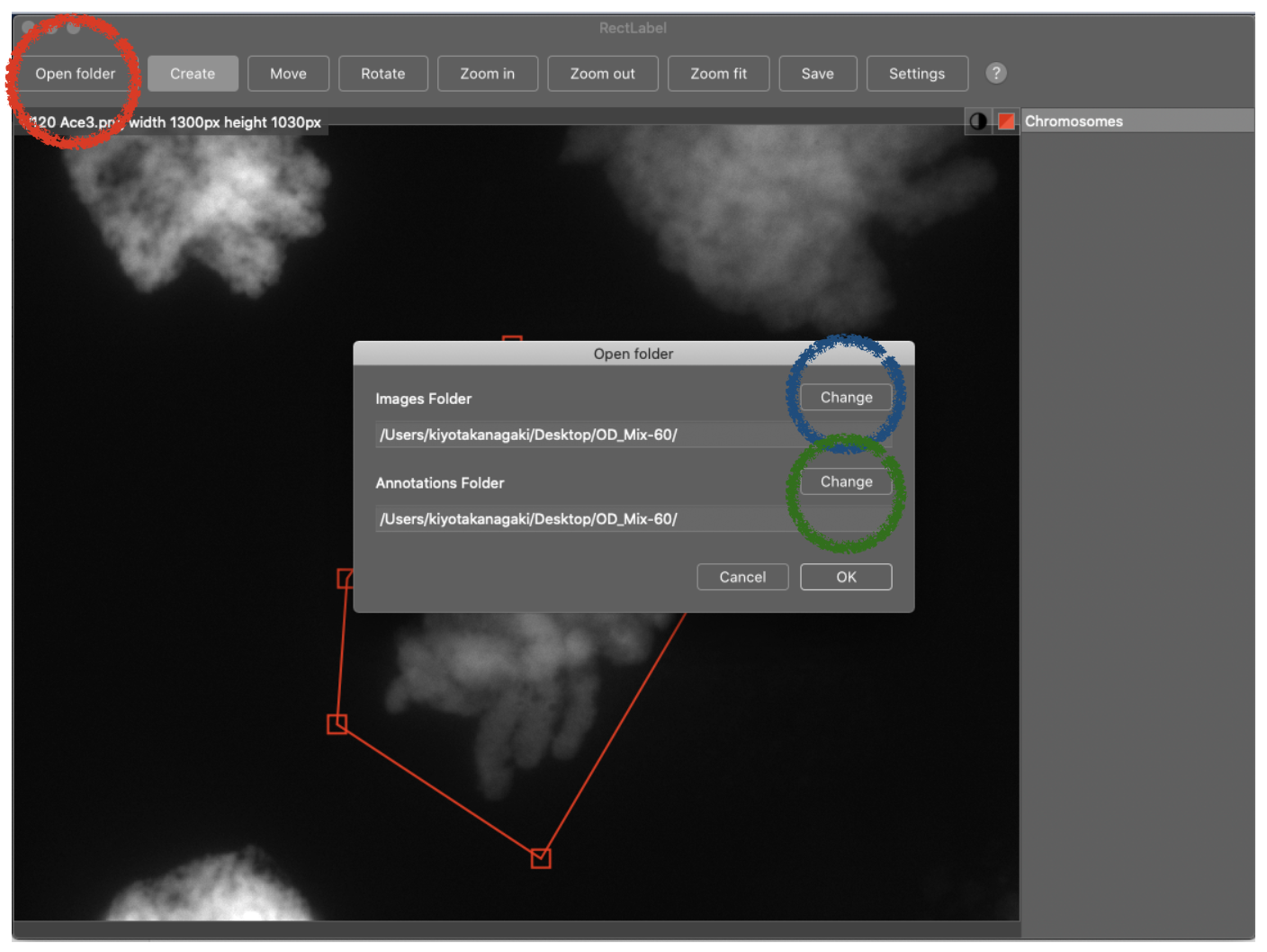
(1) RectLabelを起動し、“Open folder”ボタンをクリックしてください。開いたウインドウ内の“Change”ボタンをクリックして、アノテーションする画像の入っているフォルダとアノテーションを保存するフォルダ(今回は同じフォルダ)を選んでください。
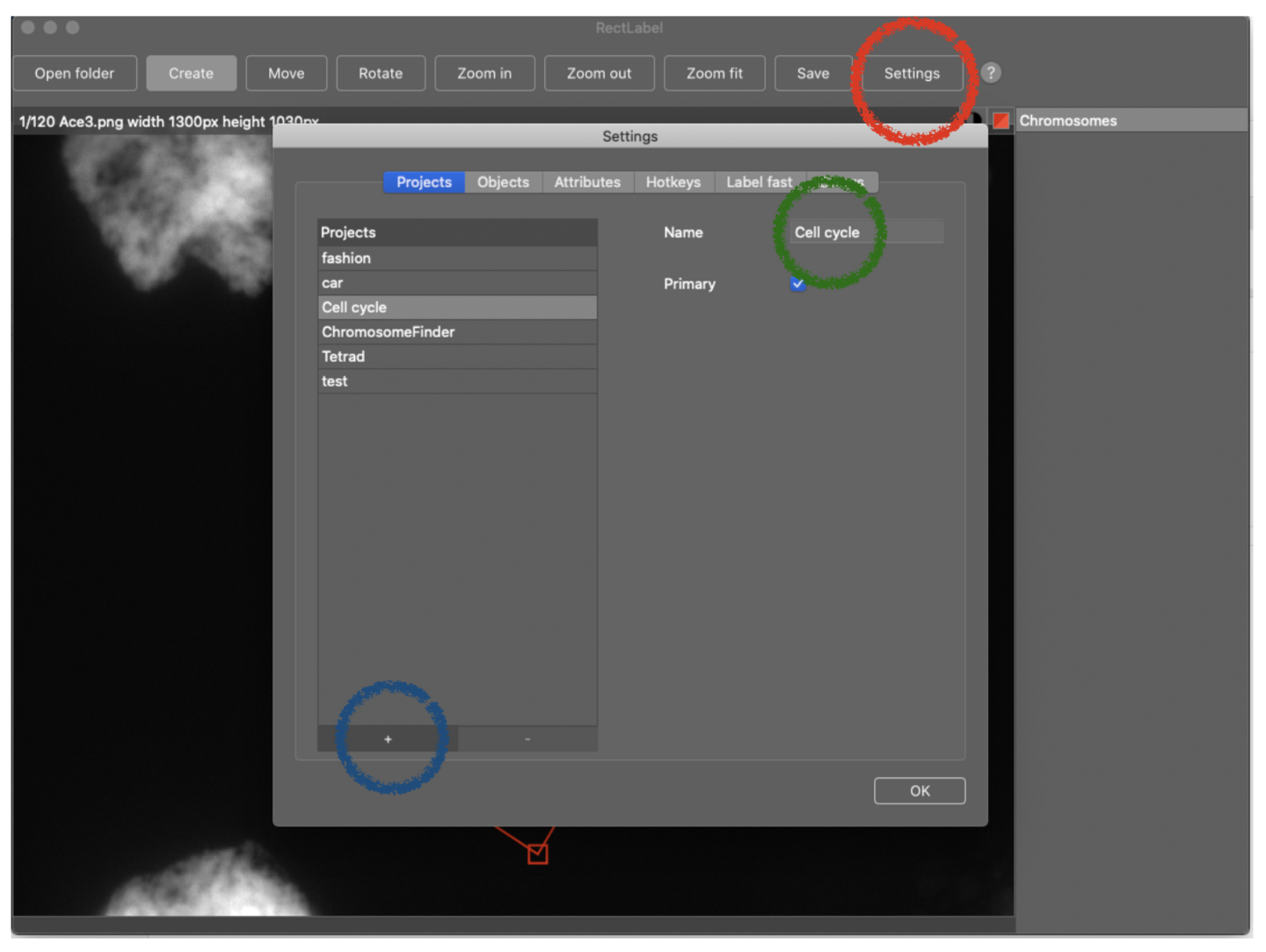
(2) “Settings”ボタンをクリックして、続いて開いたウインドウ内の“+”をクリックしてプロジェクト名を入力してください。
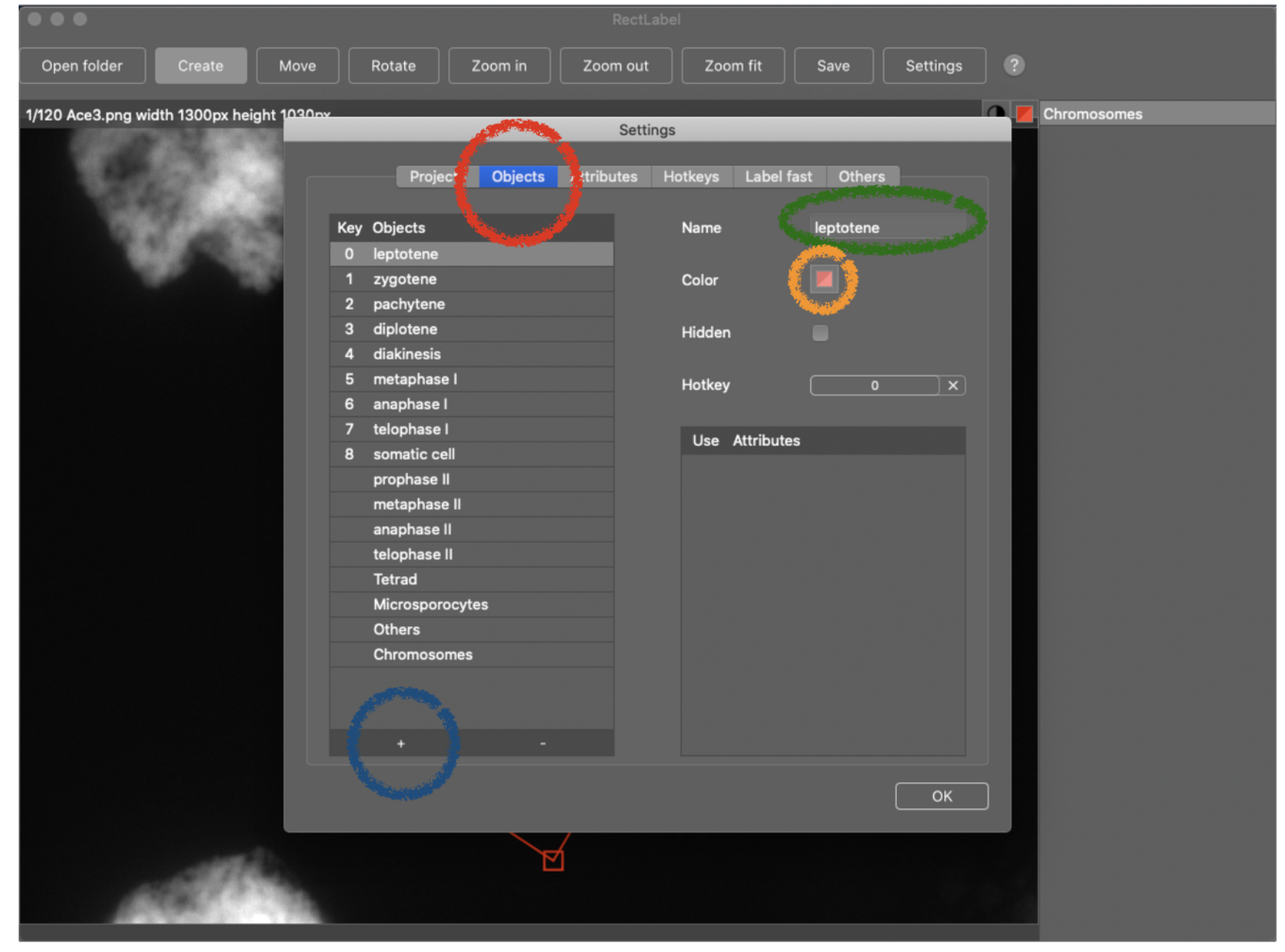
(3) “Object”ボタンをクリックし、物体を登録するために“+”ボタンをクリックしてください。続いて、物体名を入力し、それを示す枠の色を指定してください。
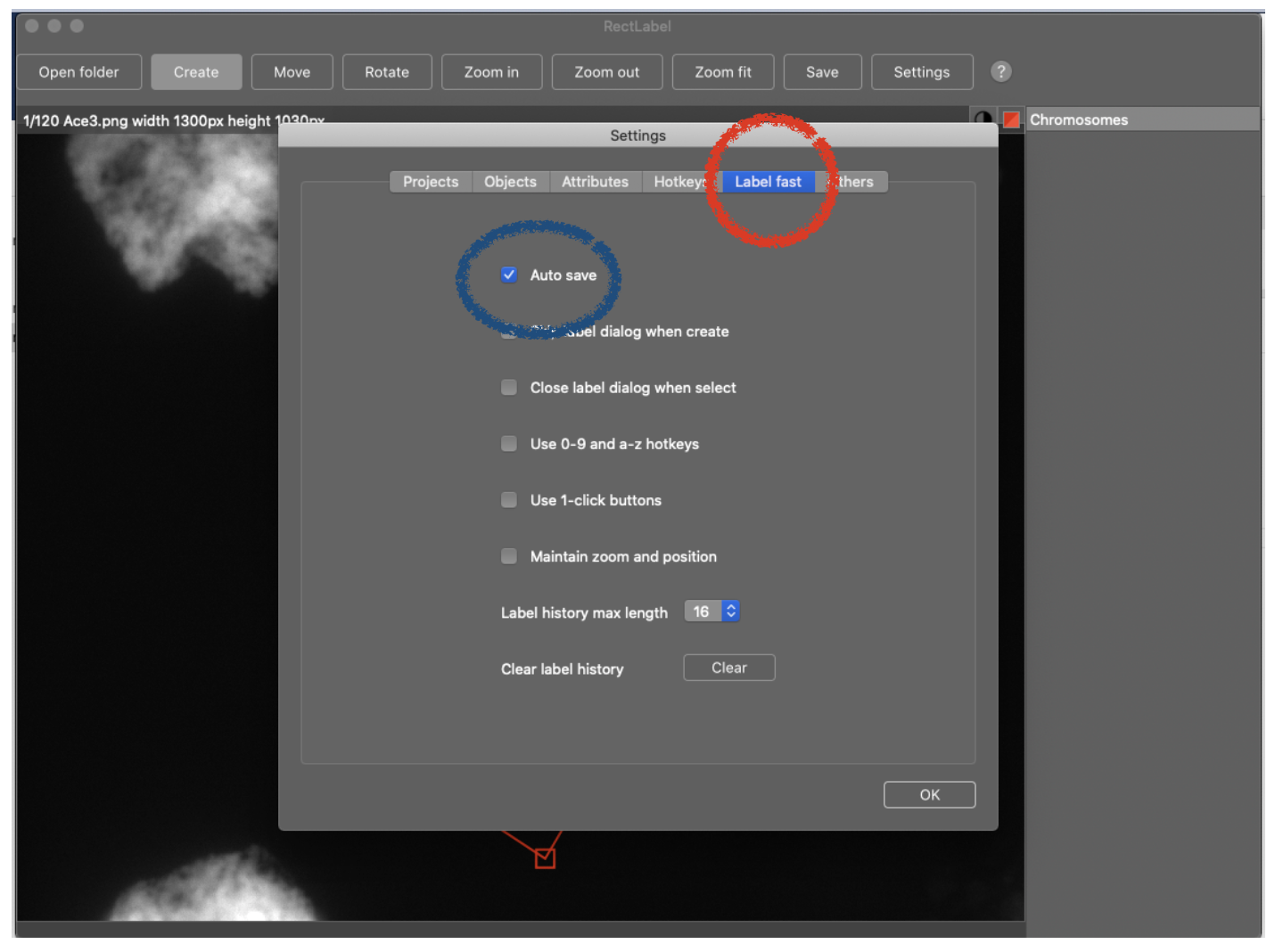
(4) “Label fast”ボタンをクリックし、“Auto save”のチェックボックスにチェックを入れてください。このオプションをオンにすることにより、画像をスクロールするごとに自動的にアノテーションが保存されます。
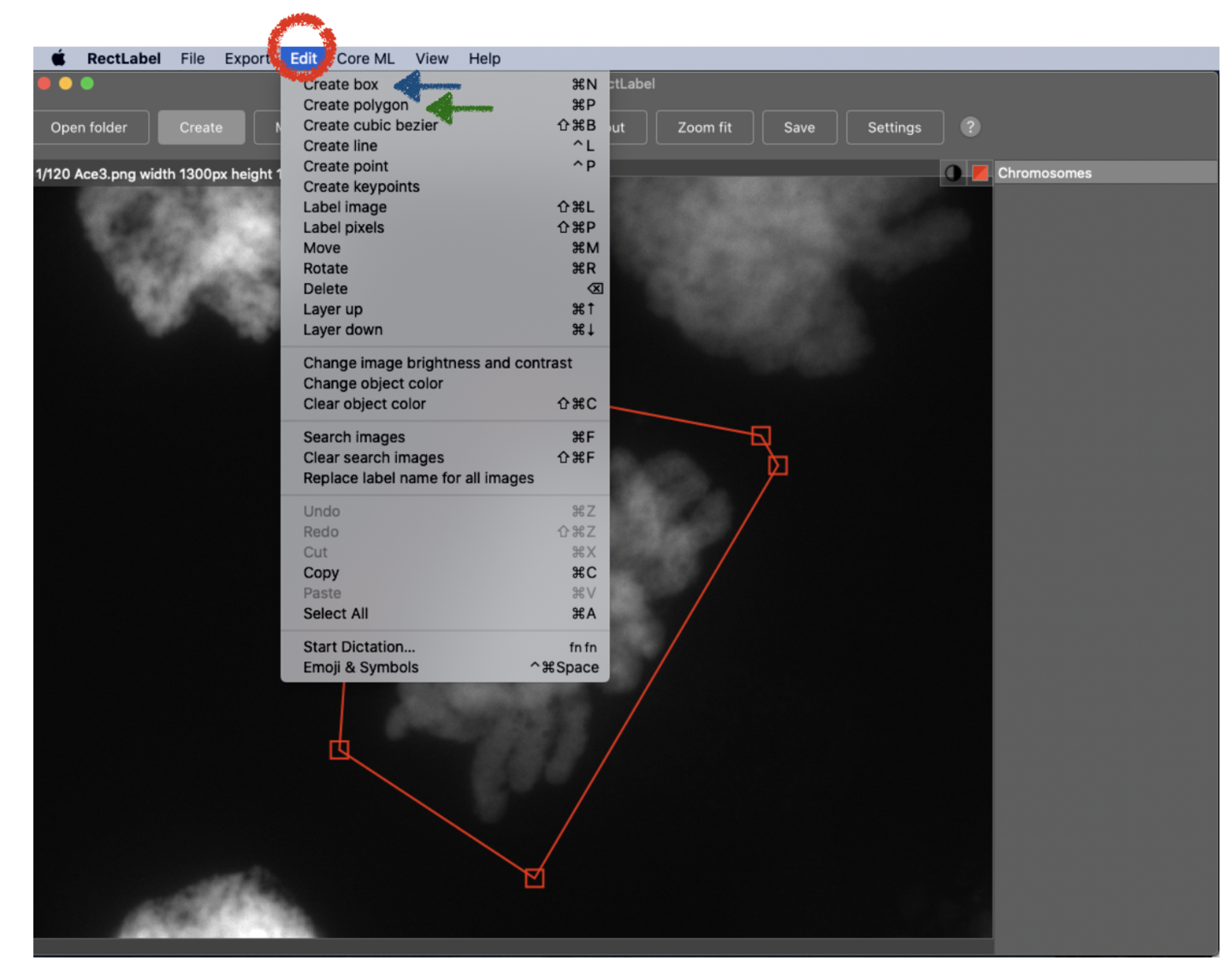
(5) “Edit”をプルダウンして、簡易的なアノテーションの場合は“Create box”を選択してください。物体をより正確に多角形を用いて選択したい場合には“Create polygon”を選択してください。
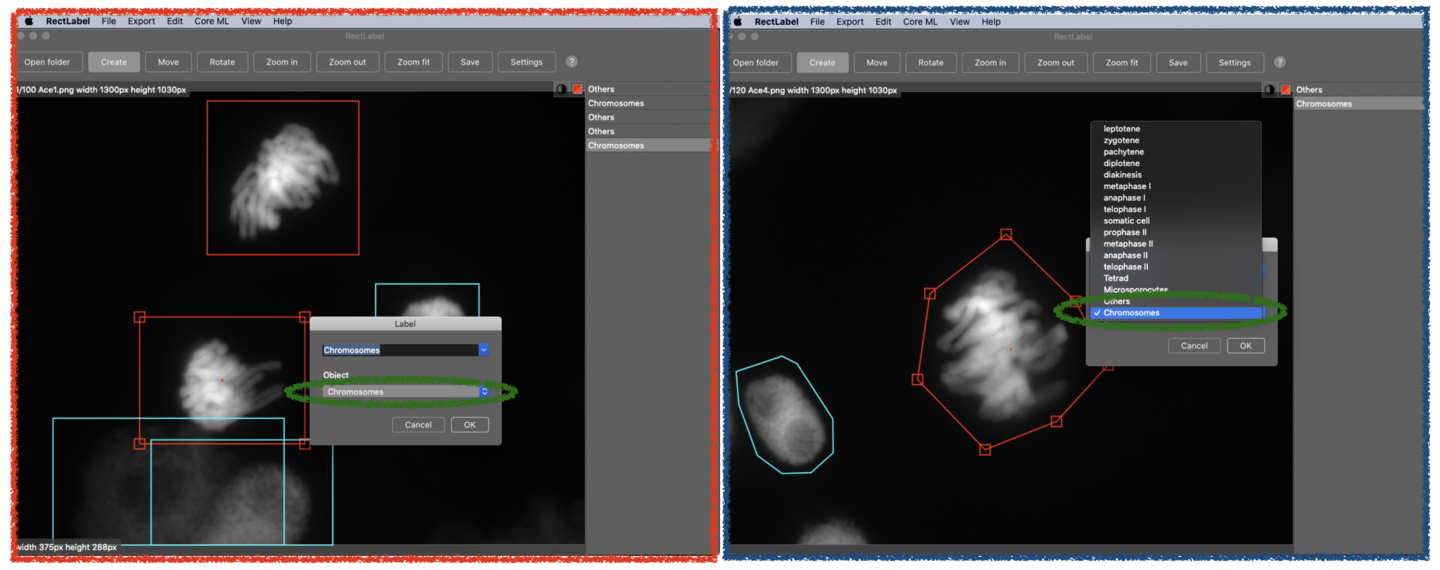
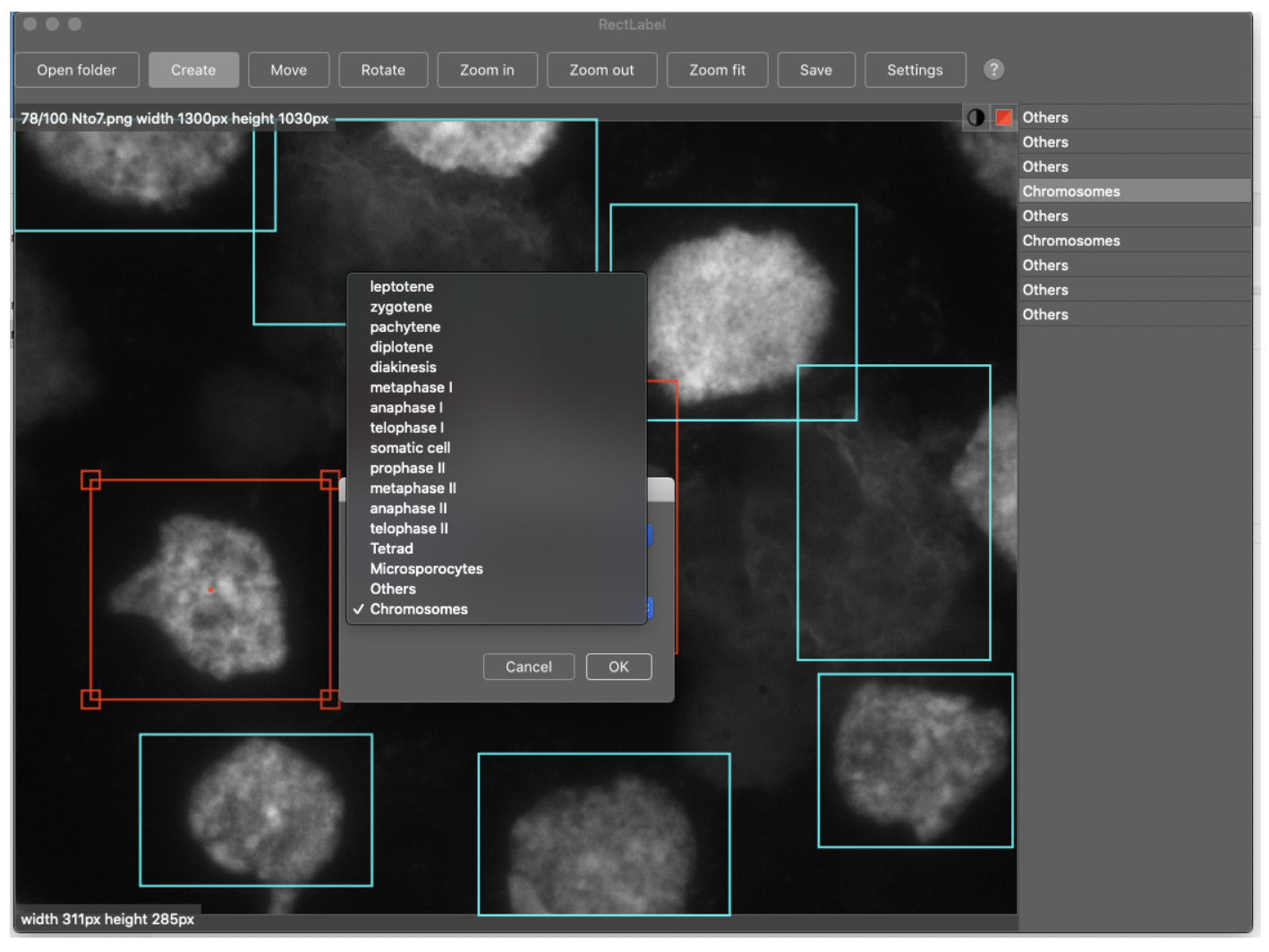
(6) 画像中の物体をアノテーションしてください。“Create box”を用いる場合は、対角にあたる2点をクリックすれば四角形を作ることができます。“Create polygon”の場合は、物体を囲うように複数の点をクリックし、起点と終点をつなぐためにリターンキーを押します。続いて“Object”をプルダウンして物体名を選択します。矢印キーの右を押すと次の画像にスクロールできます。
学習のためのアノテーション(Object detector) 裏技?
「いちいち手動で大量にアノテーションしたくない」「少しでも楽したい」という場合は、少数のアノテーションで学習させたモデルや既存のモデルを使って「AIによるアノテーション」を行い、出力された結果の範囲や名前を修正すると、少ない労力で、大量のアノテーションを行うことができます。一見関係なさそうな物体を検出するモデルでも、個々の物体の範囲だけは正確に検出することがあり、この場合は物体の名前を変更するだけで、アノテーションを完了することができます。ただし、この方法では、ボックスを用いたアノテーションになり、ポリゴンを用いて細かい範囲を指定することはできません。
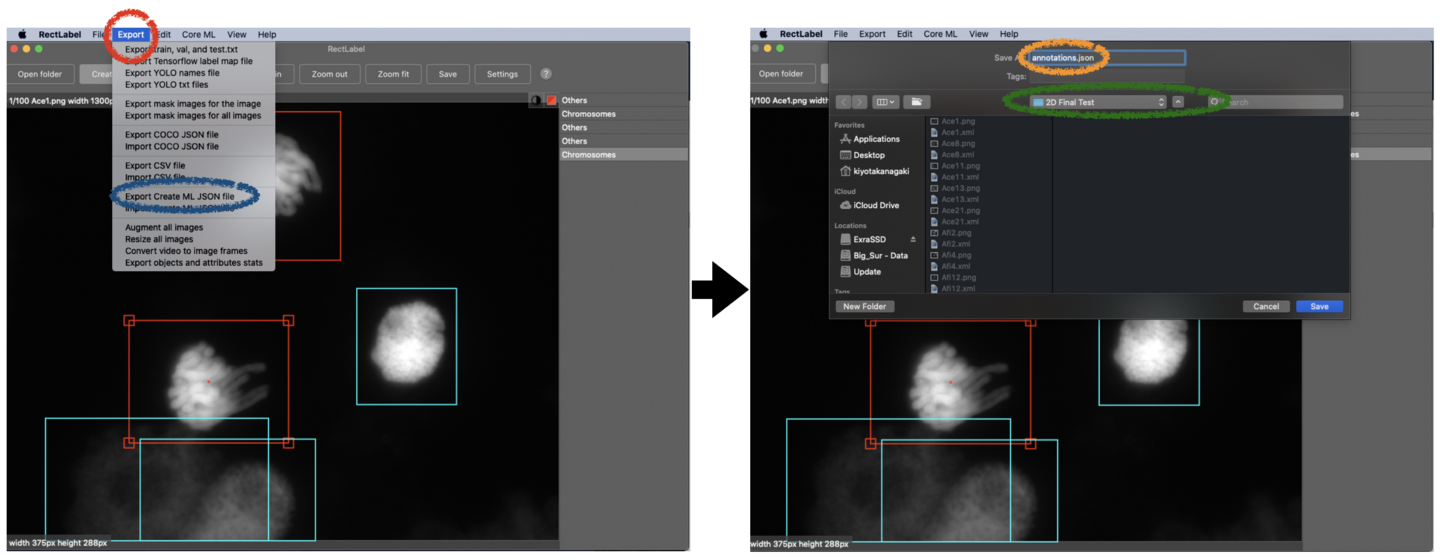
(7) 全ての画像中の物体のアノテーションが終了したら“Export”をプルダウンして“Export Create ML JSON file”を選択してください。そして、JSONファイルをアノテーションした画像と同一のフォルダに“annotations”という名前で保存してください。
Learning
学習
(1) X-codeを起動して“X-code”のプルダウンメニューから、“Open Developer Tool”内にある“Create ML”を選択してください。
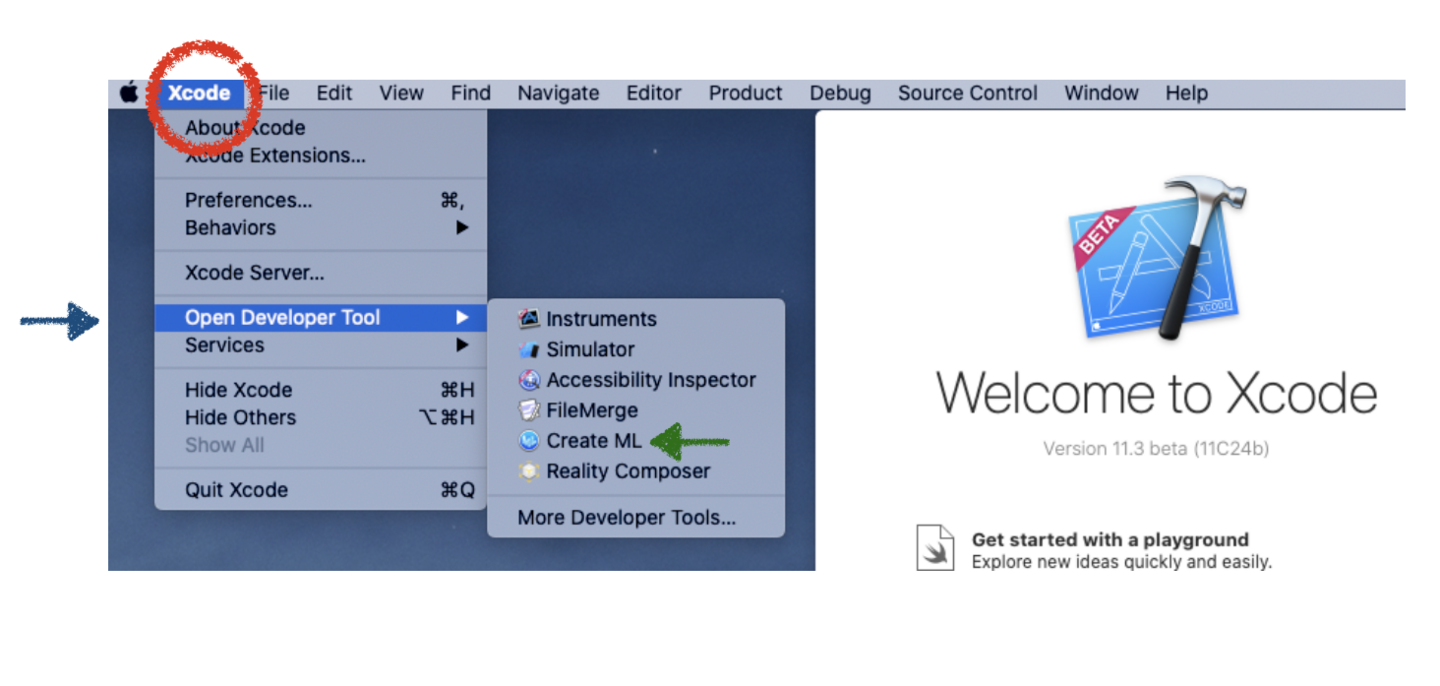
(2) “New Document”ボタンをクリックしてください。
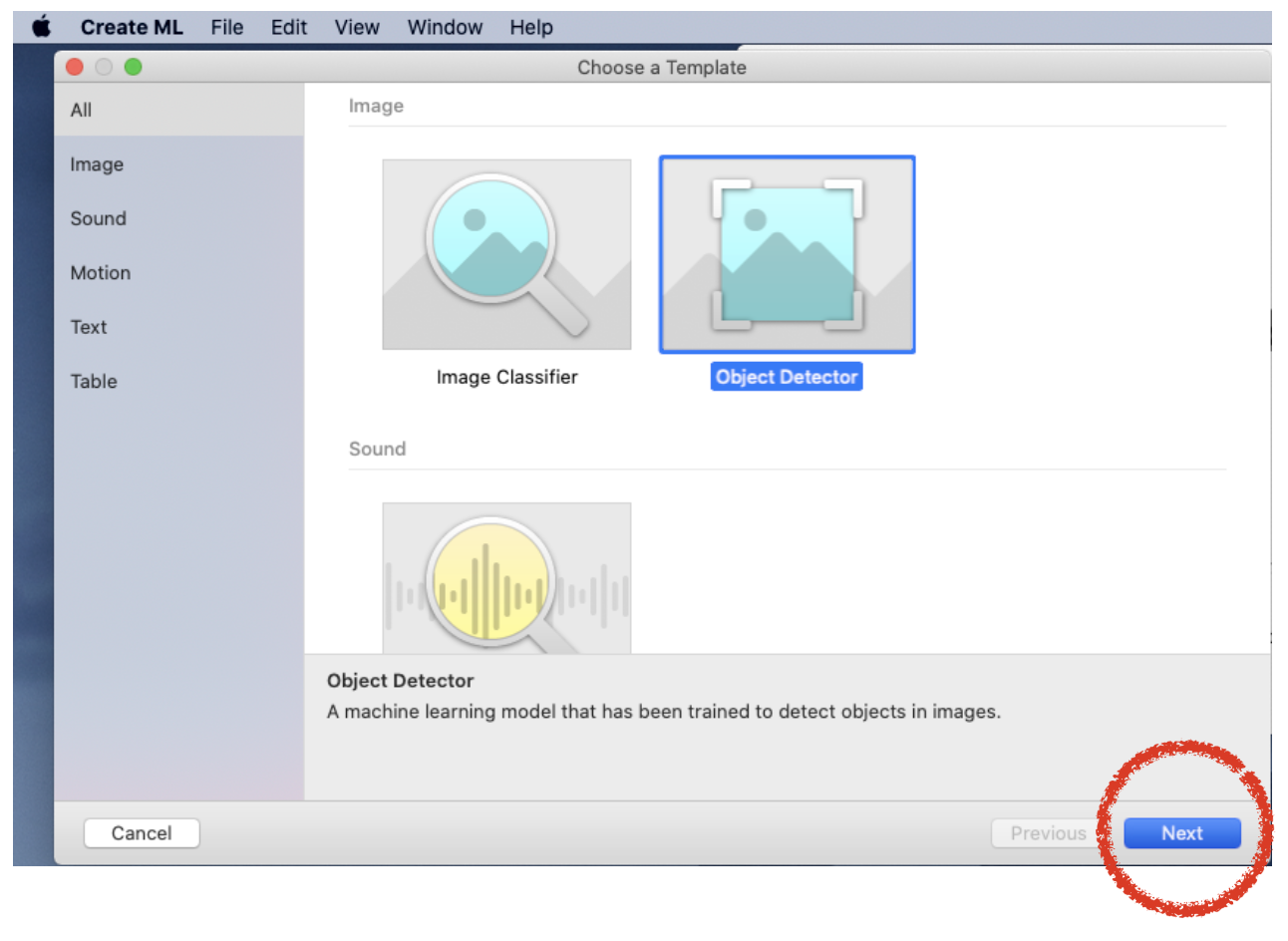
(3) “Object Detector”を選び、“Next”ボタンをクリックしてください。
(4) “Project Name”を入力し、“Next”ボタンをクリックしてください。
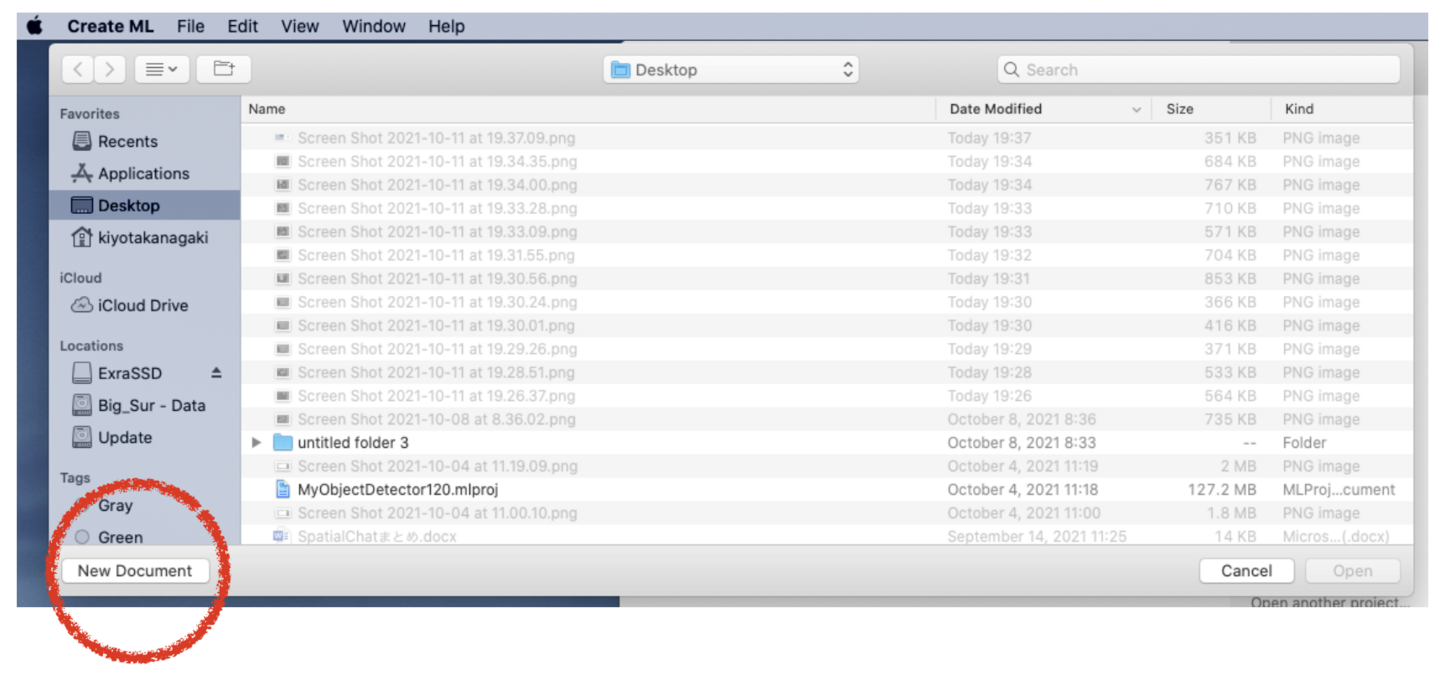
(5) project fileを作成するために“Create”ボタンをクリックしてください。
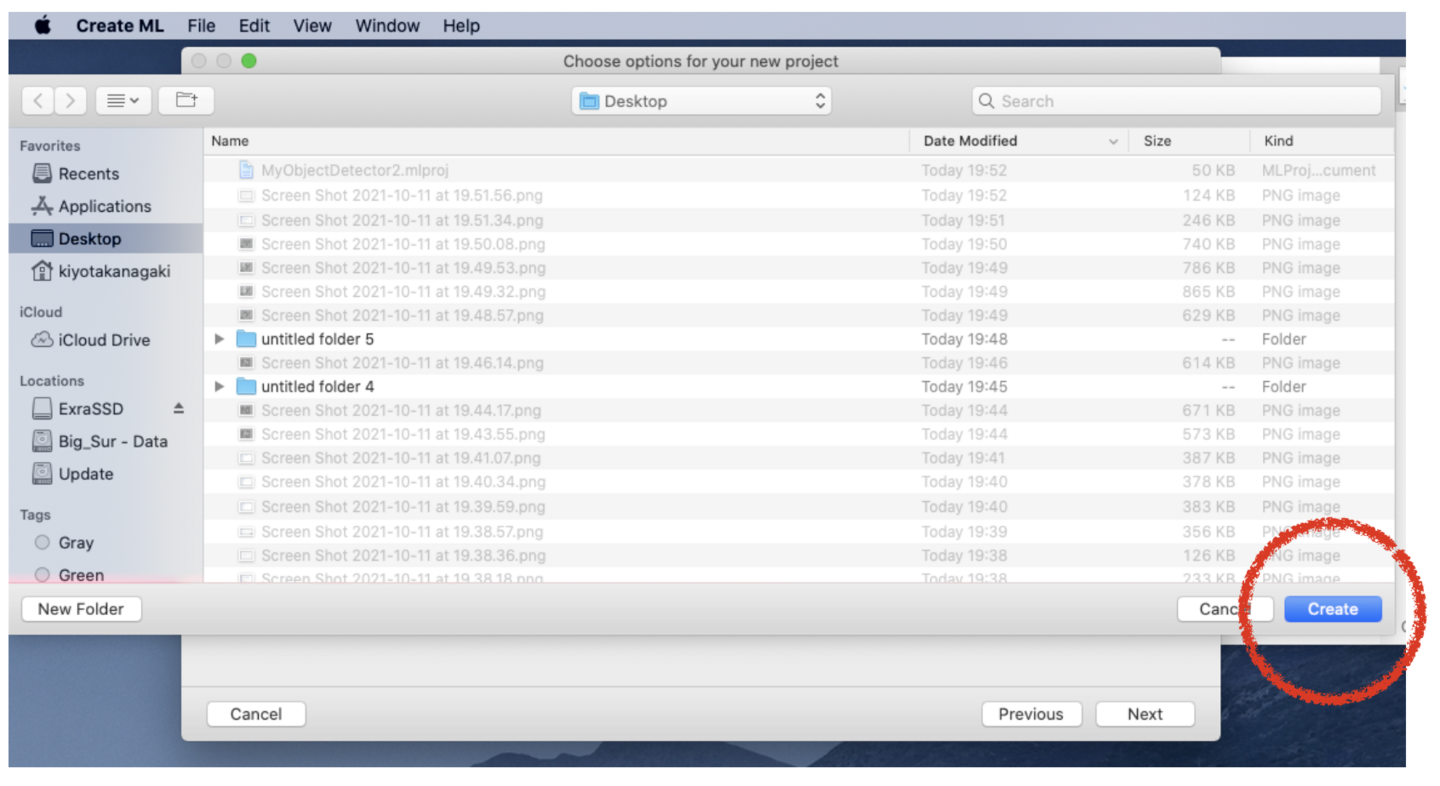
(6) “Choose”をプルダウンして、「学習のためのアノテーション」で準備した学習用の画像とjsonファイルの入ったフォルダーを選択してください。うまく読み込みができるとアイテム数とクラス数が表示されます。

オプション:
Muximum Iterationsが自動的に表示されますが、必要に応じて数を変更することができます。私の経験では、自動的に表示された数値は、適切な値でした。
In the ver 3
Create ML ver 3では、Object Detectorに大きな変更がありました。複雑な変更ですので、ver 1の学習の説明の後に変更点を説明します。
(7) “Train” ボタンをクリックして学習を開始します。
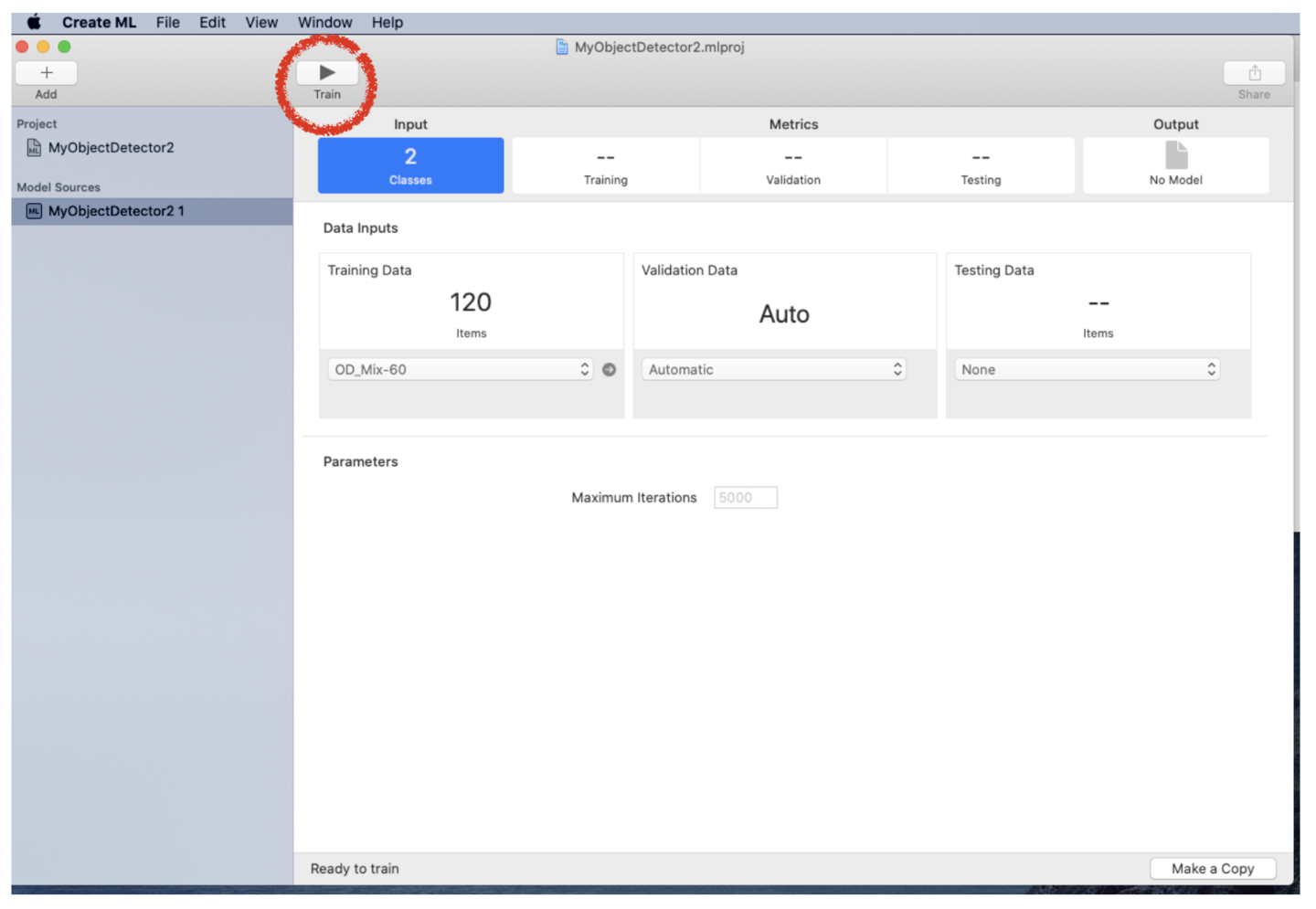
すぐに学習が始まり、多くの場合は数十分から数時間で終わります。
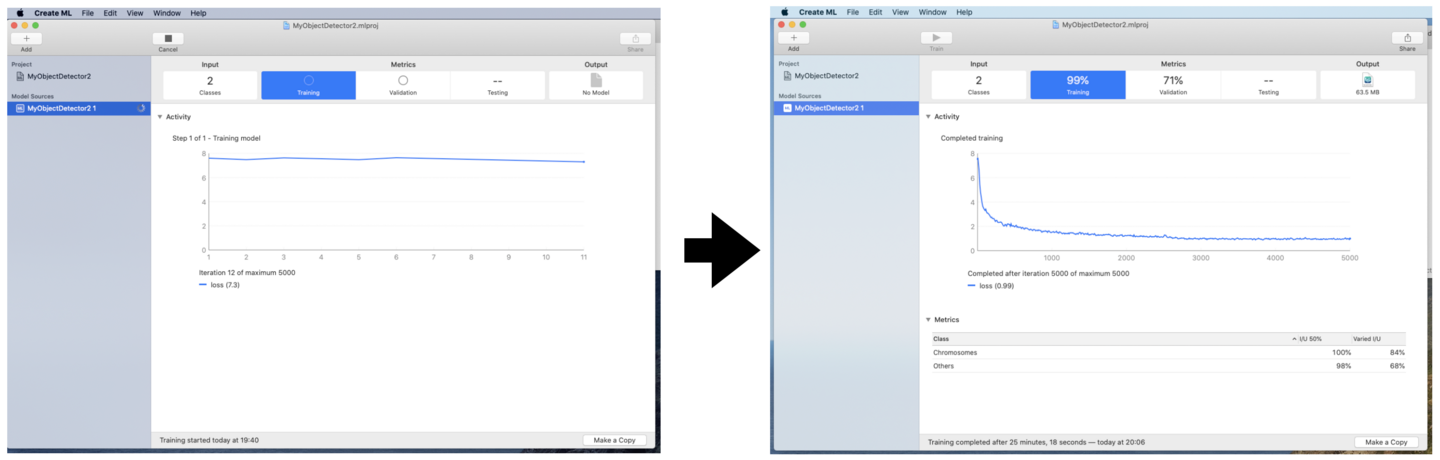
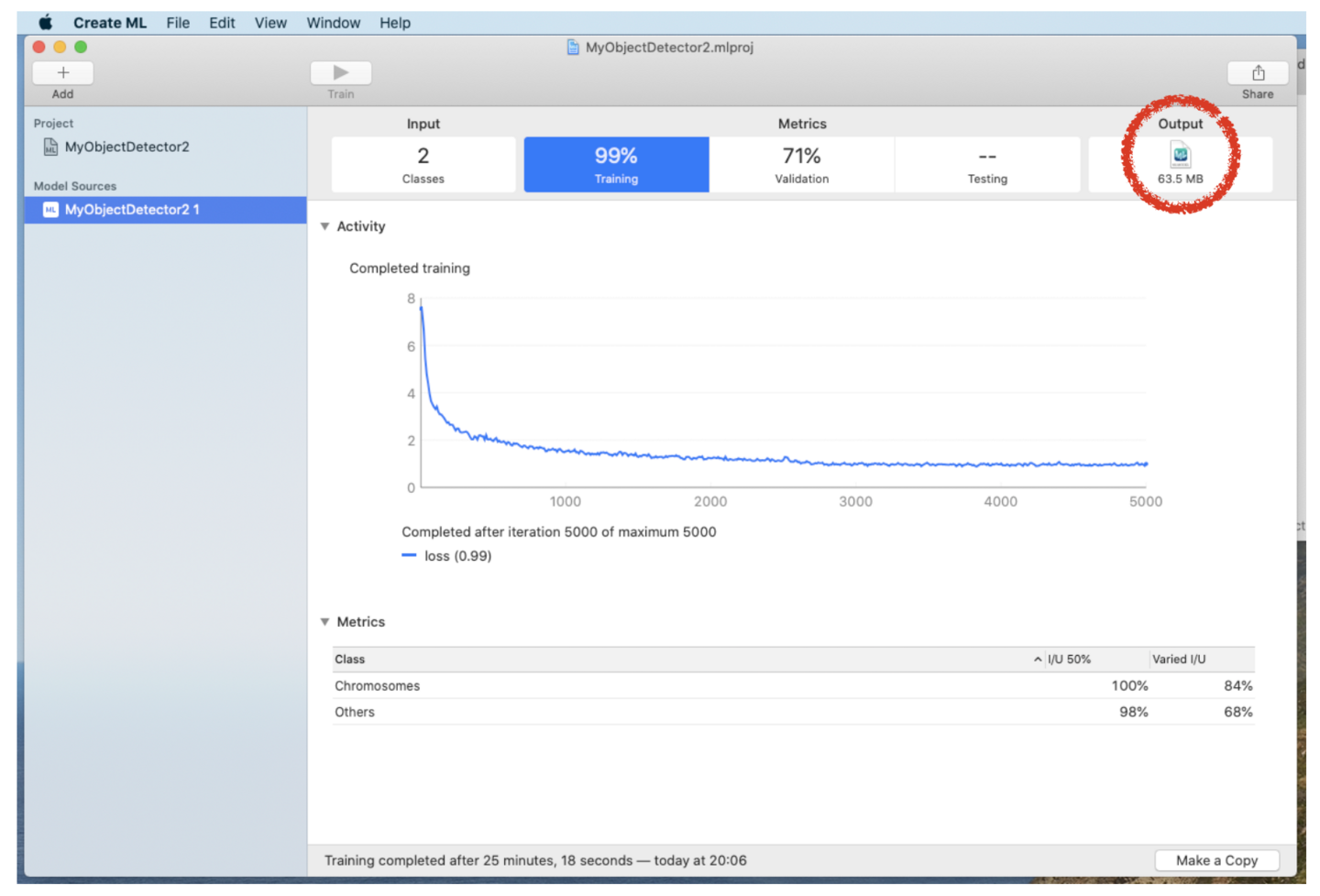
(8) 生成されたモデルを保存するには、“output” の下に表示されているファイルアイコンを保存したい場所にドラッグします。
In the ver 3
Create ML ver 3では、Object Detectorに大きな変更がありました。

In the ver 3
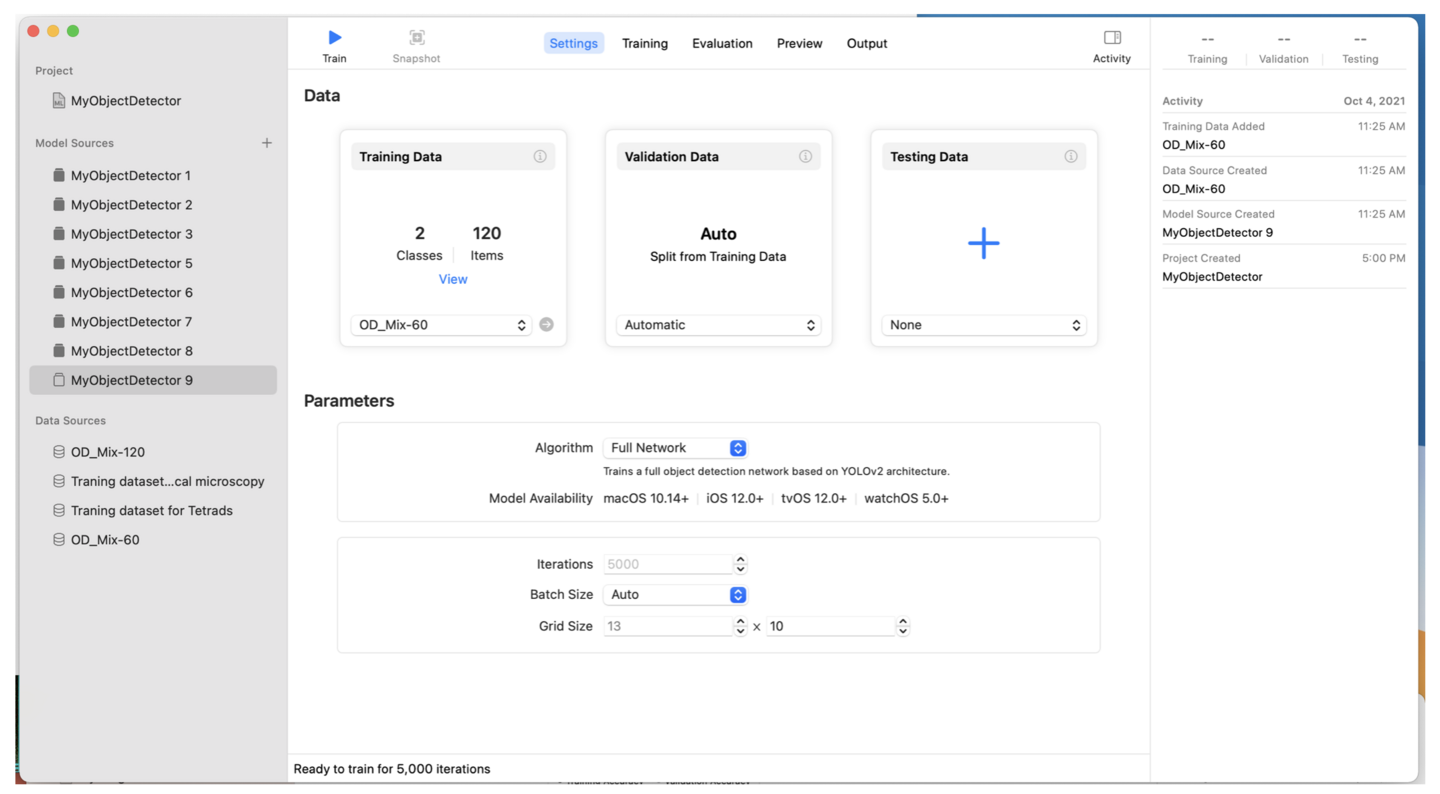
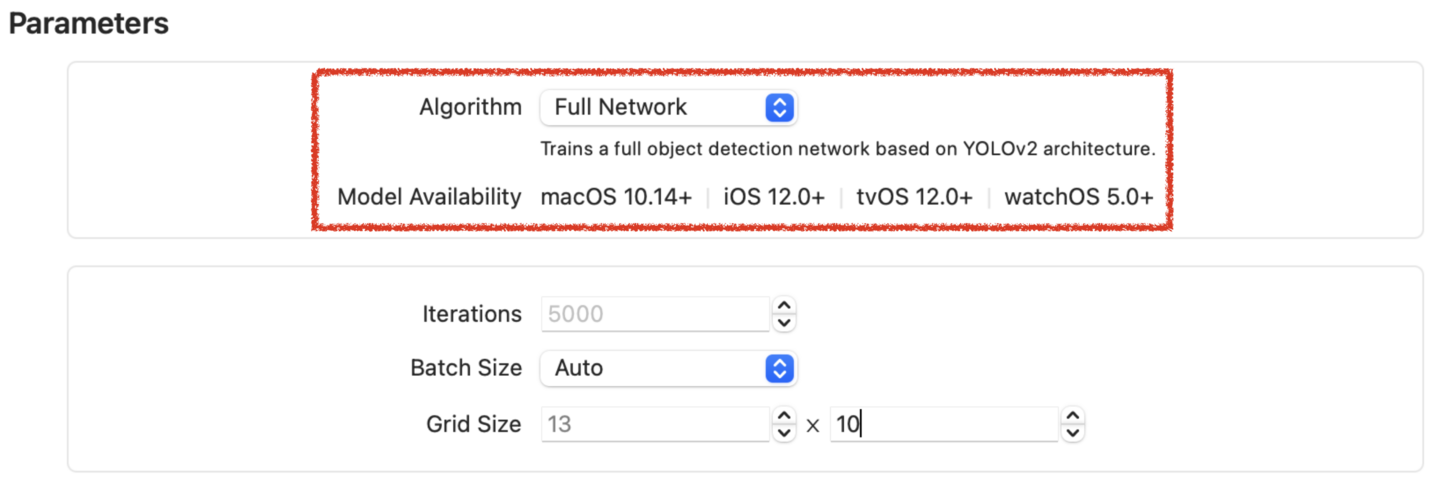
Create ML ver 3のObject DetectorではAlgorithmが選択できるようになりました。
In the ver 3
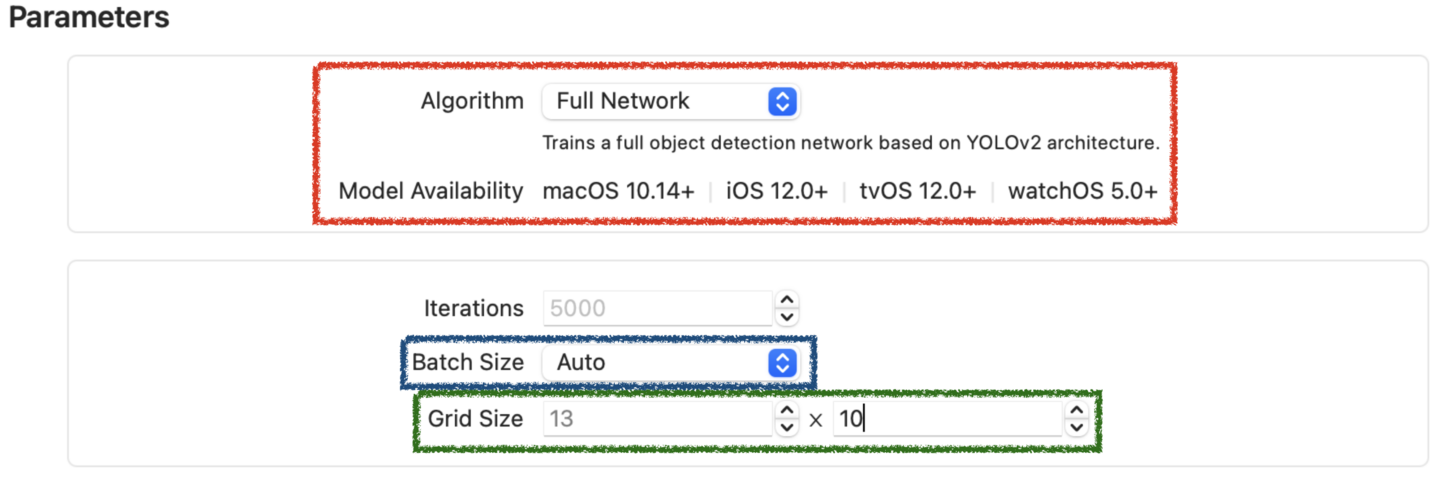
Full Networkの場合は、ver 1と類似した学習を行い、生成されたモデルはmacOS 10.14以降で使用することができます。この場合、Grid Sizeというパラメータが表示されます。
Grid Sizeは、1-30 x 1-30の範囲で選択できますので、学習画像の横x縦の比率に近い値を選択してください(例えば1300x1030の画像の場合、13x10または26x20)。この値が画像の縦横比と極端にずれると学習画像がゆがむことになり、学習結果の悪化につながります。また、理論的にはGrid Sizeの値が大きいほど近接した複数の物体を検出しやすくなりますが、Grid Sizeの値が大きいほど計算量が多くなり、学習が長時間になります。Grid Sizeの値をより大きくすれば、必ずより良いモデルができるわけではなく、画像のサイズ、画像に占める物体の割合などの条件によって結果は異なります。
Batch Sizeは、一度のiterationに使用される画像の枚数ですが、使用するコンピューターの能力に合わせて自動選択されます。この値が大きいほど学習は正確になるので、手動で変更することもできますが、選んだ値が大きすぎると学習が長時間になります。
In the ver 3
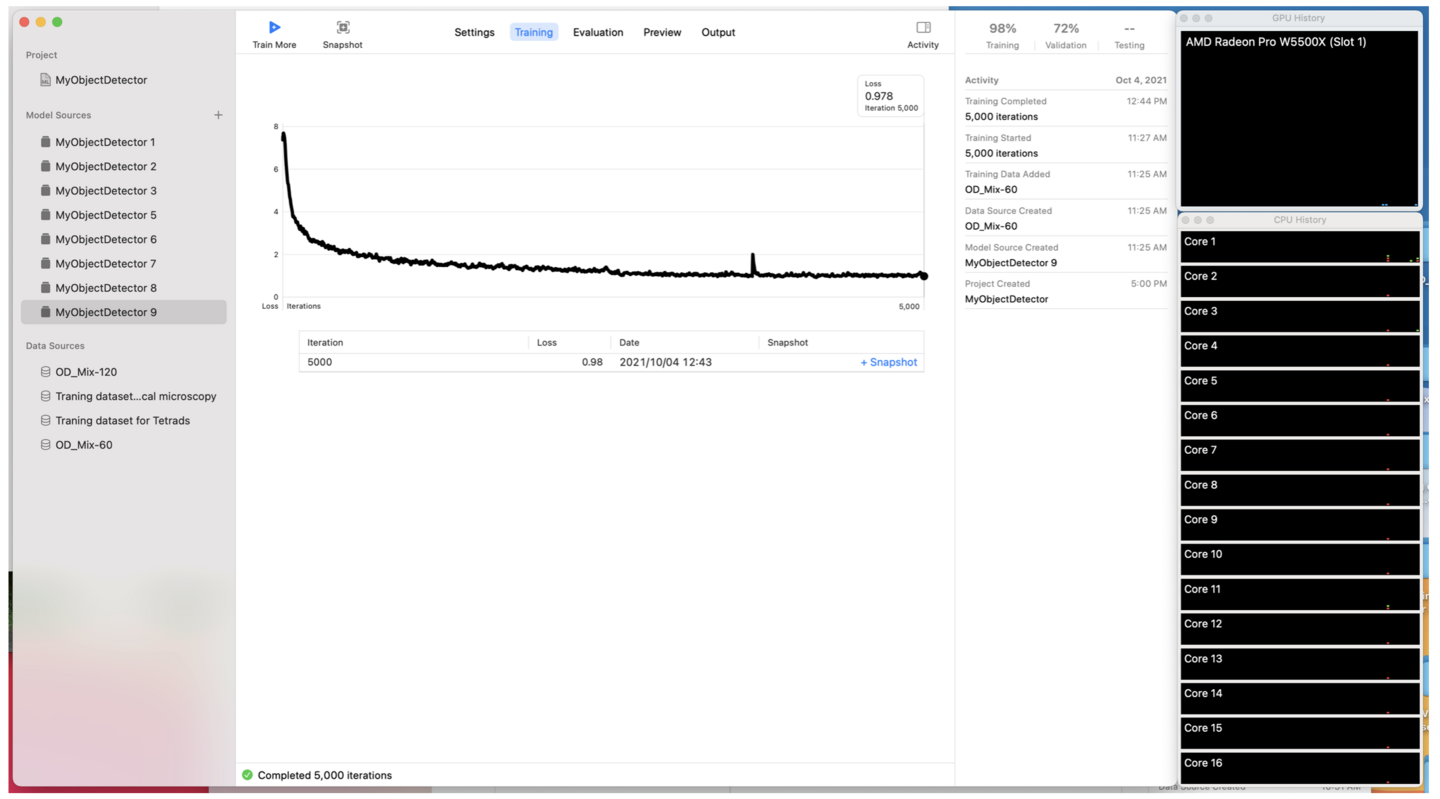
Create ML ver 3のFull Networkを用いて生成されたモデルは、Grid size 13 x 13の場合、ver 1で生成されたモデルと同等の物体検出精度を持つことが多いです。画面に占める割合が小さな物体を検出する場合は、グリッド内にその物体が複数入り込まない程度のグリッドの大きさになる様にGrid sizeに入力する値を変更すると検出精度が向上します。しかし、グリッドの大きさを小さくするために「Grid size」に大きな値を入力すると(入力する値とグリッドの大きさが反対の関係なのでややこしいですね...)学習時間が増加します。また、使用するGPUやMacによっては設定できる値の上限が異なります(正確には設定できないわけではなく、すぐにエラーが出る。実際のGPUと使用可能なGrid sizeの情報はコチラを参考にしてください)。たとえ、エラーなく学習させることができても、学習時間が膨大になることもあります。ですので、まずは短時間で学習が可能なGrid size 13 x 13を試してみて、良いモデルができなければ、受容できる学習時間の範囲でGrid sizeの値を大きくしていくのが現実的かと思います。
In the ver 3
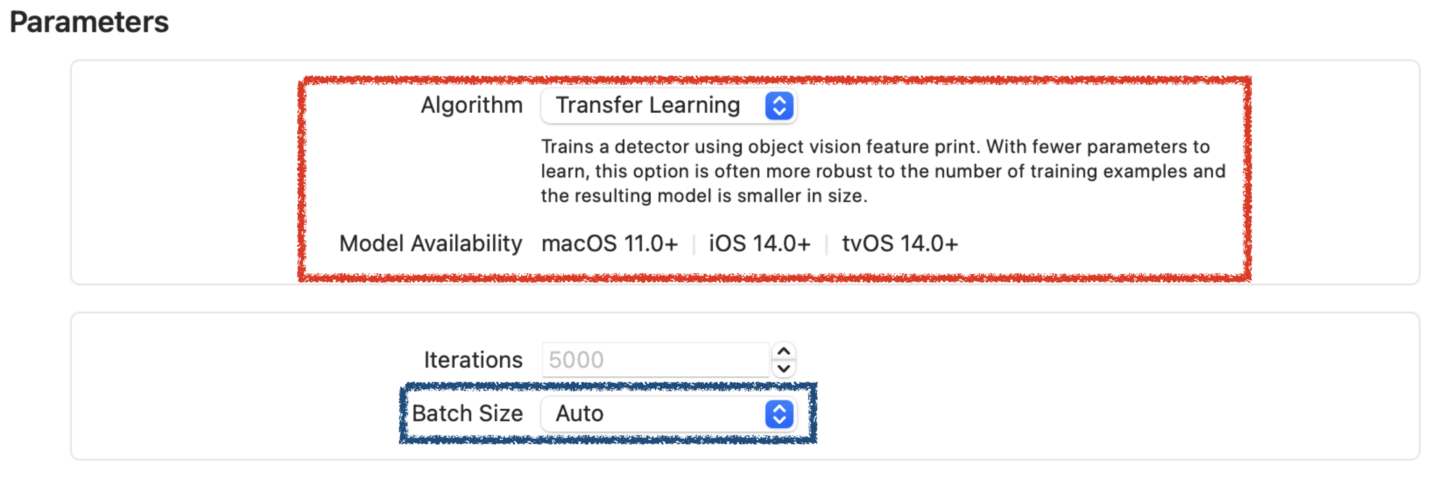
Transfer Learningで生成されたモデルはmacOS 11以降でしか使用できません。この学習では、Macの写真アプリで物体検出に使われているモデルを利用して転移学習を行うので、学習に必要な物体数は各クラス80個以上とFull Networkの200個以上に比べて少ない画像で学習することができます(いずれもアップル社の推奨値であり、これら以下でも学習は可能です)。Batch Sizeは、一度のiterationに使用される画像の枚数ですが、使用するコンピューターの能力に合わせて自動選択されます。この値が大きいほど学習は正確になるので、手動で変更することもできますが、選んだ値が大きすぎると学習が長時間になります。
In the ver 3
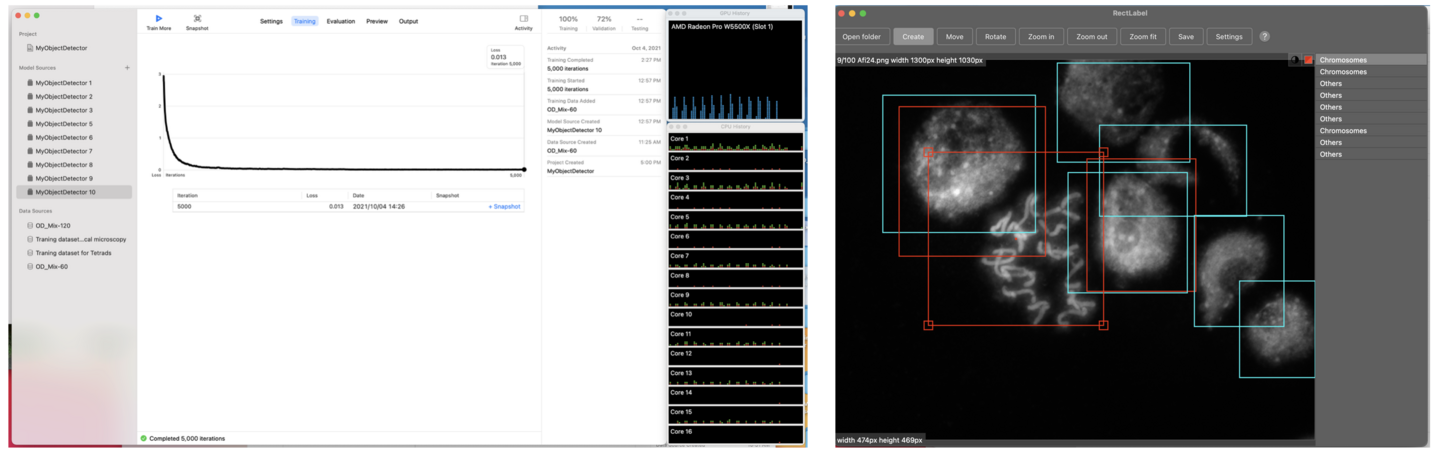
Create ML ver 3のTransfer Learningを用いて生成されたモデルは、非常に小さいloss値を示しますが、染色体データの場合、RectLabelで確認すると1つの細胞を複数回検出する間違いが多くなっています。おそらくは、写真アプリのモデルが細胞の検出に向いていないためだと思われますので、検出する物体によって、転移学習の効果が異なる可能性があります。
In the ver 3
Create ML ver 3のObject Detectionでは、ver 1とは異なるモデルが出力されるので、双方のバージョンを用いてモデル生成すれば、目的にあったモデルが生成される可能性が高まるかもしれません。
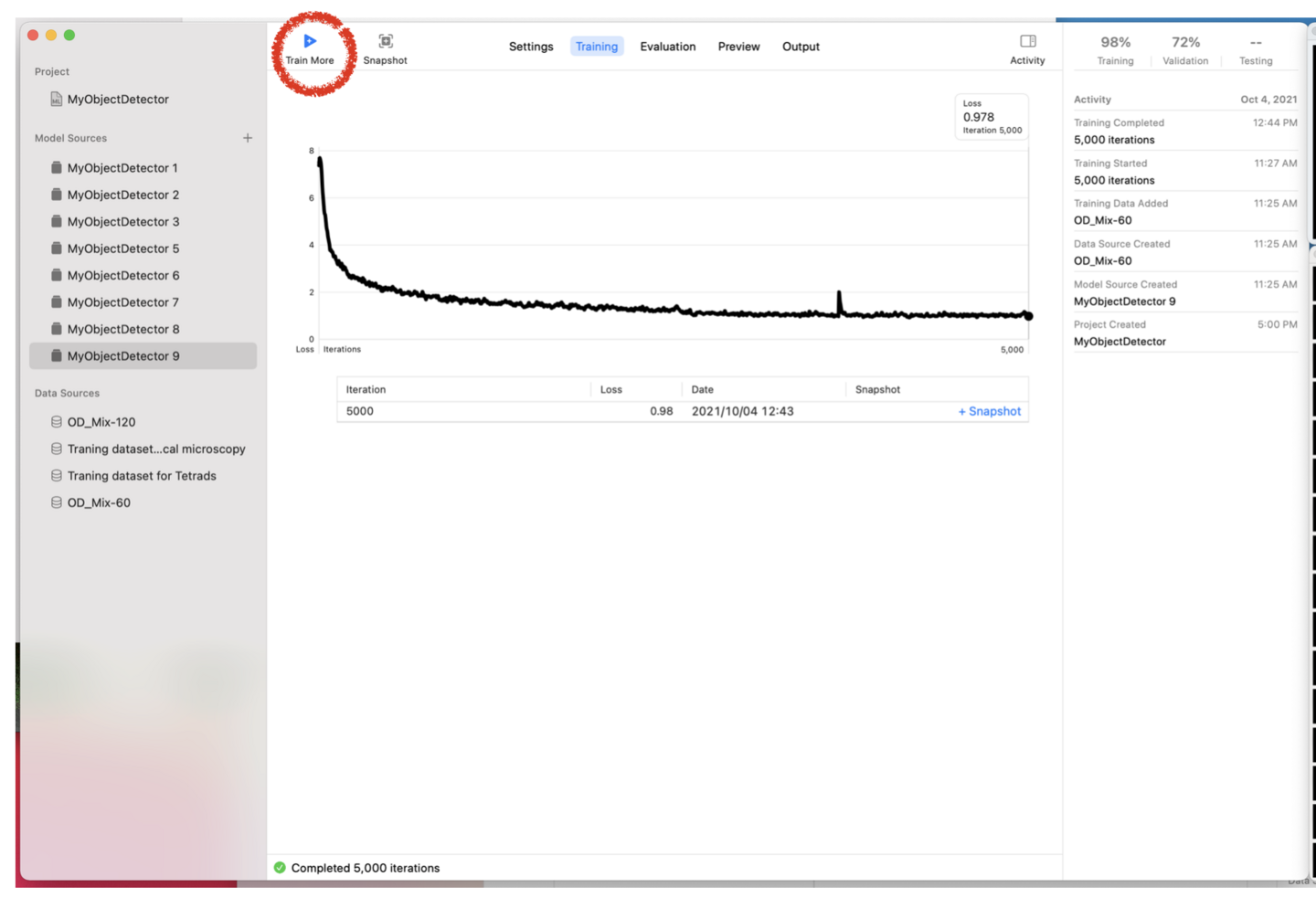
これまでに述べなかったver 3独自の機能として、「追加学習が容易」という点があります。追加学習を行う場合(Iteration回数を増やす場合)、ver 1では最初からやり直す必要がありましたが、ver 3ではTrainingウィンドウ左上に表示される「Train More」というボタンを押せば、追加学習が可能になっています。
Annotation by AI
AIによるアノテーション
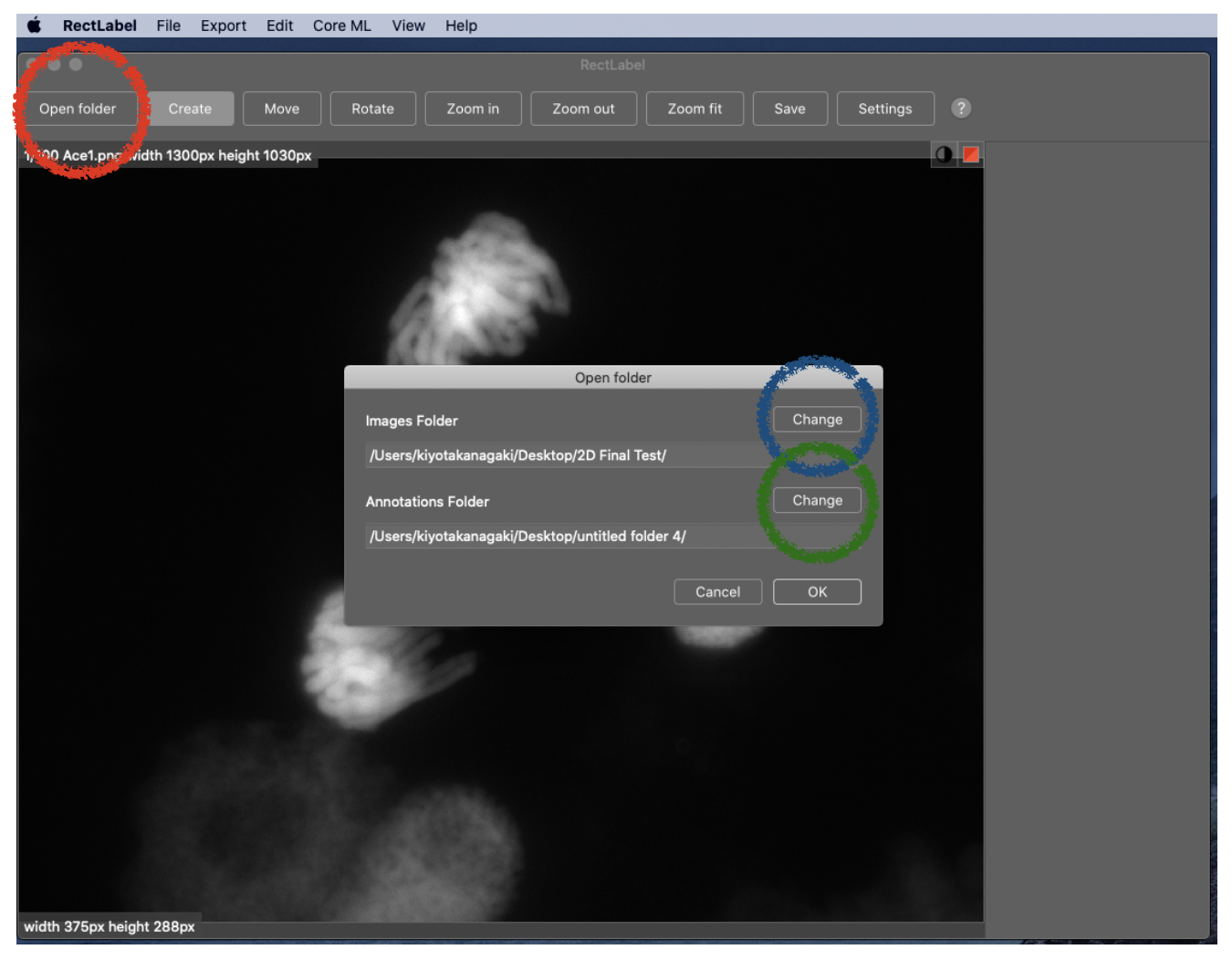
RectLabelを起動し、“Open folder”ボタンをクリックしてください。開いたウインドウ内の“Change”ボタンをクリックして、アノテーションする画像の入っているフォルダとアノテーションを保存するフォルダを選んでください。
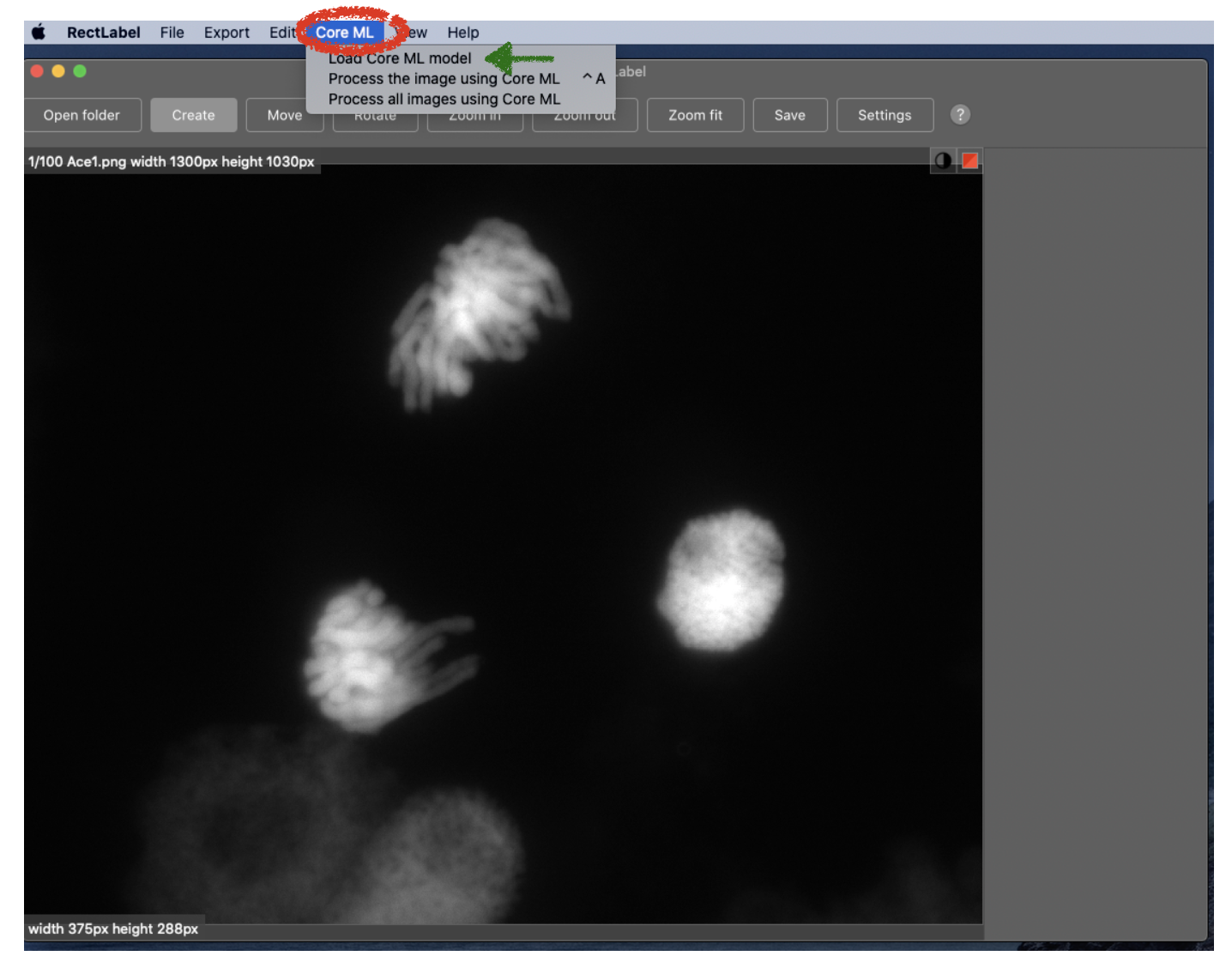
(2) “Core ML”をプルダウンして、“Load Core ML model”を選択してください。
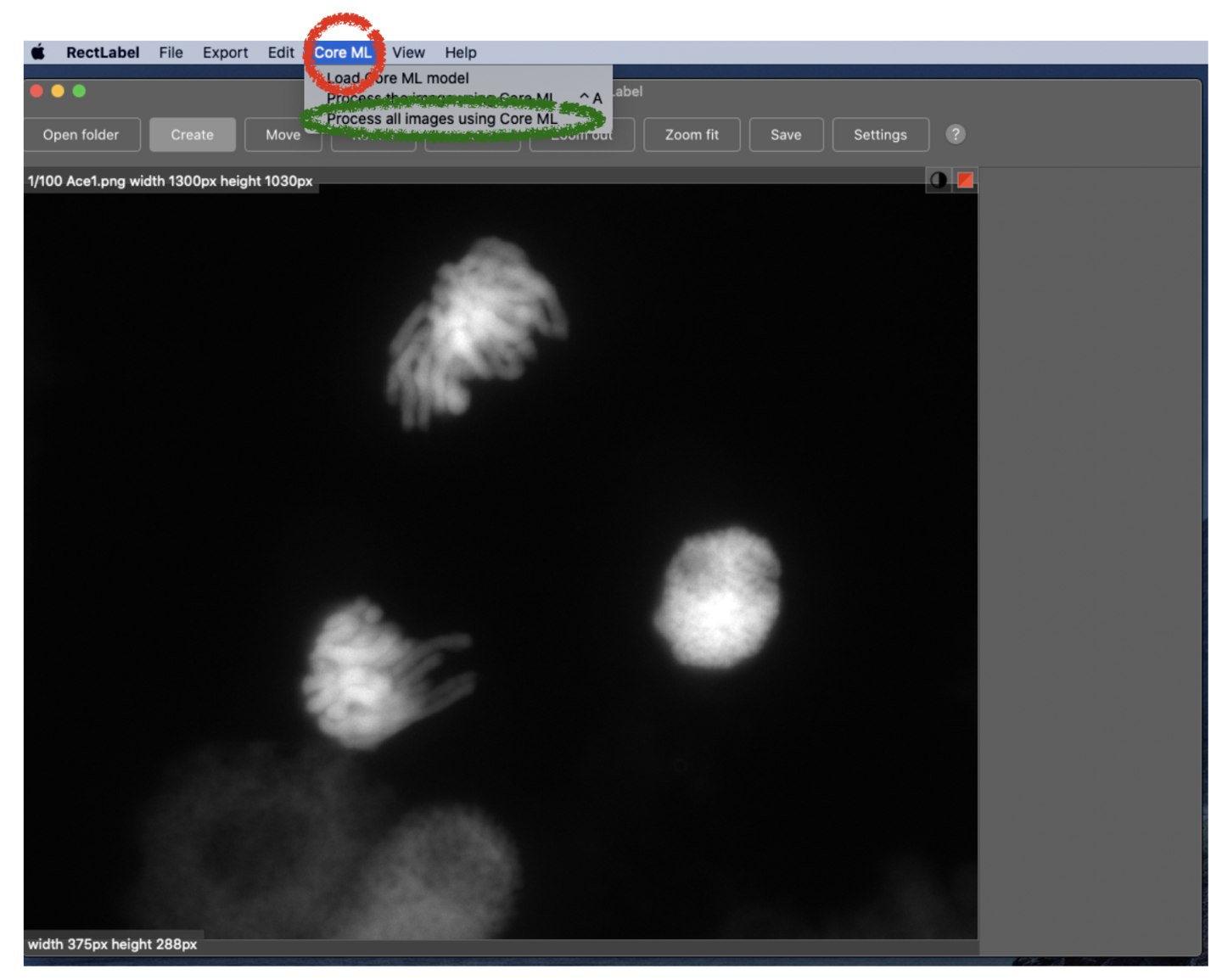
(3) “Core ML”をプルダウンして、アノテーションするために“Process all images using Core ML”を選んでください。
(4) アノテーションが終了すると、個々の物体のアノテーションが右のウインドウに表示されます。 矢印キーの右を押すと次の画像にスクロールできます。
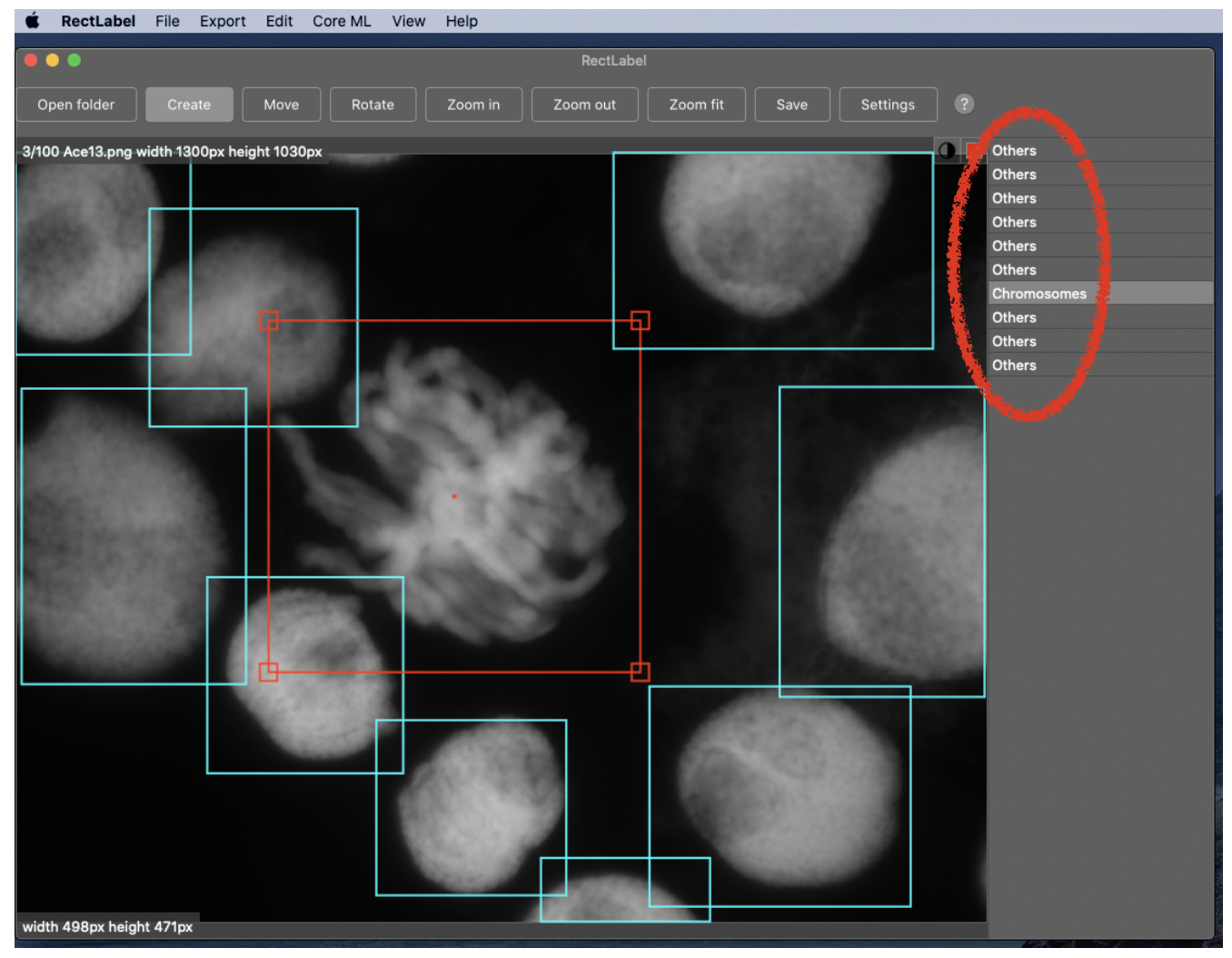
Counting Detected Objects
Object detectorモデルを用いて検出された物体のカウント
(1) ダウンロードされたCutSort-mainフォルダから“CutSort.app”の右クリックによりウインドウを表示し、続いて“Open”を選択し、開いたウインドウの“Open”ボタンをクリックしてください。
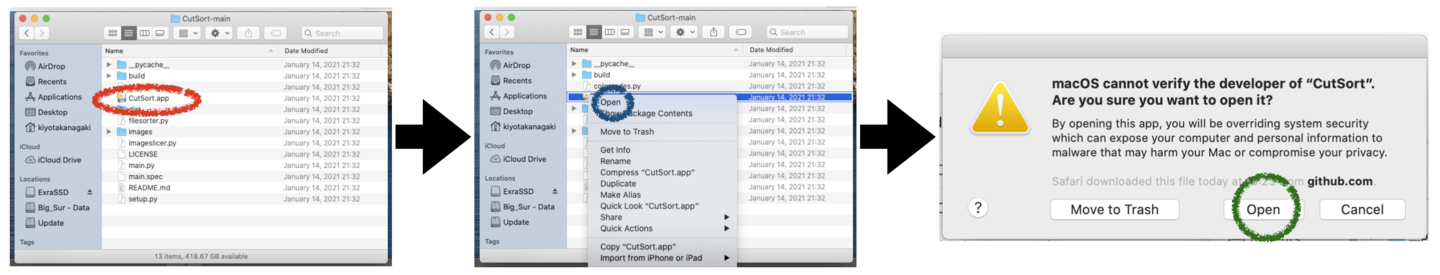
(2) “File Sorter”ボタンをクリックして、“Object detection on single images”を選択してください。

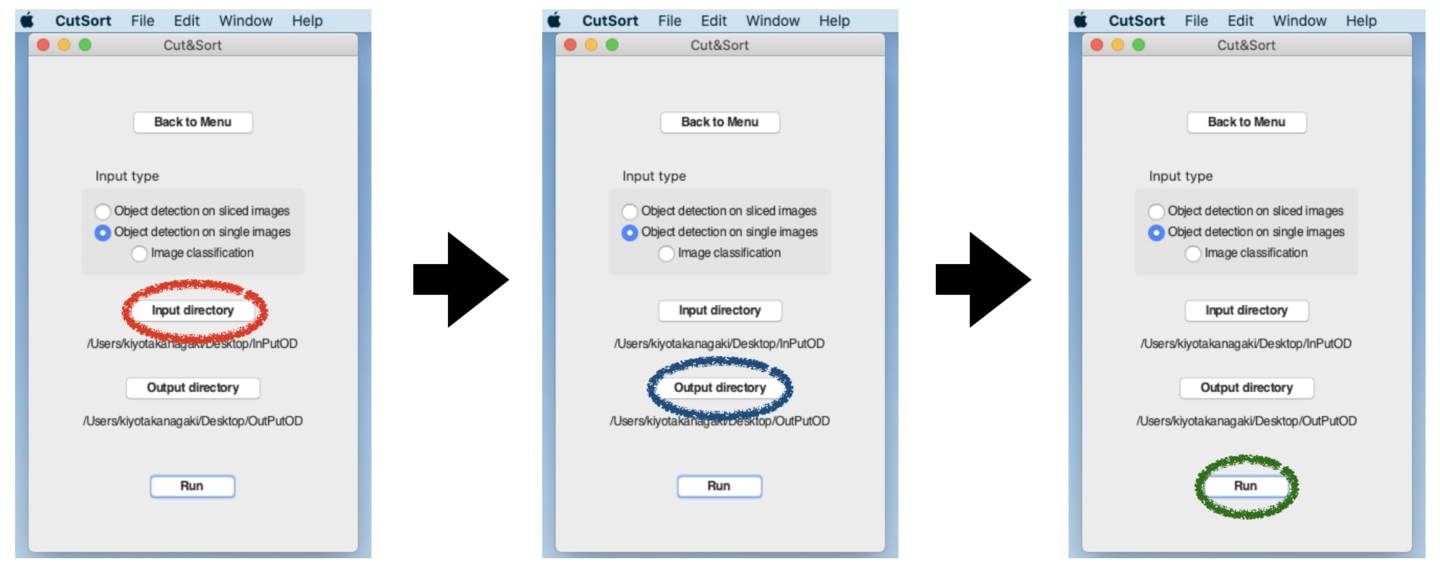
(3) RectLabelによって出力された画像と.xmlファイルを含むフォルダーを“Input directory”ボタンをクリックして選択してください。続いて、出力先のフォルダーを“Output directory”ボタンをクリックして指定してください。最後に“Run”ボタンをクリックすると画像中の物体がカウントされます。
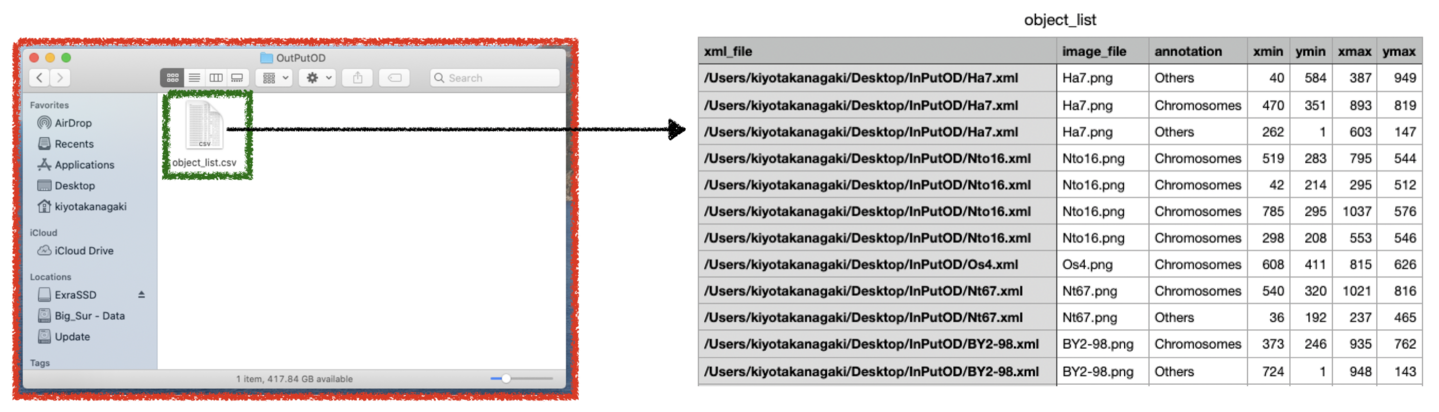
(4) 結果として、指定された出力フォルダには画像中の物体に関する情報が記載された“object_list.csv”ファイルが生成されます。このファイルには検出した物体がリストアップされていますので、エクセル等の表計算ソフトを使って全ての画像中に存在する各クラスに分類された物体の数を知ることができます。
Models
モデル
シェアされているObject detector用のモデルです。
ダウンロードして、自動検出に、ご利用ください。
さらに、みなさんの作成したモデルもご提供いただければ、順次こちらからダウンロードできる様にします。
Machine learning is
Power for the people, by the people, of the people!
1. 細胞、染色体、四分子の顕微鏡画像の検出モデル
報告された論文
Kiyotaka Nagaki, Tomoyuki Furuta, Naoki Yamaji, Daichi Kuniyoshi, Megumi Ishihara, Yuji Kishima, Minoru Murata, Atsushi Hoshino and Hirotomo Takatsuka. Effectiveness of Create ML in microscopy image classifications: A simple and inexpensive deep learning pipeline for non-data scientists. Chromosome Research in press, 2021. DOI:https://doi.org/10.1007/s10577-021-09676-z
Mix-60_OD.mlmodel
12種由来の分裂細胞を含む画像(Chromosomes: 各5枚)と4種由来の分裂細胞を含まない画像(Others: 各15枚)を元に生成したObject detector用のモデル。
Mix-120_OD.mlmodel
12種由来の分裂細胞を含む画像(Chromosomes: 各10枚)と4種由来の分裂細胞を含まない画像(Others: 各30枚)を元に生成したObject detector用のモデル。
Mix-767_OD.mlmodel
12種由来の分裂細胞を含む画像(Chromosomes: 全767枚)と4種由来の分裂細胞を含まない画像(Others: 全232枚)を元に生成したObject detector用のモデル。
Nt-120_OD.mlmodel
タバコの分裂細胞を含む画像(Chromosomes: 全120枚)と4種由来の分裂細胞を含まない画像(Others: 各30枚)を元に生成したObject detector用のモデル。
BY-2-120_OD.mlmodel
BY-2の分裂細胞を含む画像(Chromosomes: 全120枚)と4種由来の分裂細胞を含まない画像(Others: 各30枚)を元に生成したObject detector用のモデル。
Ha-120_OD.mlmodel
ヒマワリの分裂細胞を含む画像(Chromosomes: 全120枚)と4種由来の分裂細胞を含まない画像(Others: 各30枚)を元に生成したObject detector用のモデル。
OD_Mix-767+Keyence-150.mlmodel
12種由来の分裂細胞を含む画像(Chromosomes: 全767枚)と4種由来の分裂細胞を含まない画像(Others: 全232枚)にKeyence顕微鏡で撮影した分裂細胞を含む画像150枚を加えたデータを元に生成したObject detector用のモデル。
Confocal.mlmodel
ニンニクの根の共焦点顕微鏡画像中のinterphase(873個)、prophase(202個)、 metaphase(119個)、anaphase(41個)、telophase(109個)を元に生成したObject detector用のモデル。
Tetrads.mlmodel
イネ の四分子画像292個を元に生成したObject detector用のモデル。
Annotated Images for Learning
学習用アノテーション済み画像ファイル
モデル生成に使った学習用アノテーション済み画像ファイルです。
圧縮ファイルを解凍して生成されたフォルダをそのままCreate MLで読み込めますので、システムの動作確認や練習にご利用ください。
また、これらの画像に違うアノテーションをつければ、転移学習用にも使用可能です。
加えて、これらの画像にご自身で用意した画像を付け加えて学習することにより、少量の画像をご自身で用意するだけで、ご自身で用意した画像だけで学習するよりも高精度のモデルを作ることも可能です。
さらに、みなさんの作成した学習用アノテーション済み画像データもご提供いただければ、順次こちらからダウンロードできる様にします。
Machine learning is
Power for the people, by the people, of the people!
1. 細胞、染色体、四分子の顕微鏡画像
これらの画像が使用された論文
Kiyotaka Nagaki, Tomoyuki Furuta, Naoki Yamaji, Daichi Kuniyoshi, Megumi Ishihara, Yuji Kishima, Minoru Murata, Atsushi Hoshino and Hirotomo Takatsuka. Effectiveness of Create ML in microscopy image classifications: A simple and inexpensive deep learning pipeline for non-data scientists. Chromosome Research in press, 2021. DOI:https://doi.org/10.1007/s10577-021-09676-z
OD_Mix-60.zip
Object detector用の12種由来の分裂細胞を含む画像(Chromosomes: 各5枚)と4種由来の分裂細胞を含まない画像(Others: 各15枚)とjasonファイル。
OD_Mix-120.zip
Object detector用の12種由来の分裂細胞を含む画像(Chromosomes: 各10枚)と4種由来の分裂細胞を含まない画像(Others: 各30枚)とjasonファイル。
OD_Mix-767.zip
Object detector用の12種由来の分裂細胞を含む画像(Chromosomes: 全767枚)と4種由来の分裂細胞を含まない画像(Others: 全232枚)とjsonファイル。
OD_Nt-120.zip
Object detector用のタバコの分裂細胞を含む画像(Chromosomes: 全120枚)と4種由来の分裂細胞を含まない画像(Others: 各30枚)とjsonファイル。
OD_BY-2.zip
Object detector用のBY-2の分裂細胞を含む画像(Chromosomes: 全120枚)と4種由来の分裂細胞を含まない画像(Others: 各30枚)とjsonファイル。
OD_Ha-120.zip
Object detector用のヒマワリの分裂細胞を含む画像(Chromosomes: 全120枚)と4種由来の分裂細胞を含まない画像(Others: 各30枚)とjsonファイル。
OD_Mix-767+Keyence-150.zip
Object detector用の12種由来の分裂細胞を含む画像(Chromosomes: 全767枚)と4種由来の分裂細胞を含まない画像(Others: 全232枚)にKeyence顕微鏡で撮影した分裂細胞を含む画像150枚を加えたデータとjsonファイル。
Confocal.zip
Object detector用のニンニクの根の共焦点顕微鏡画像中のinterphase(873個)、prophase(202個)、 metaphase(119個)、anaphase(41個)、telophase(109個)とjsonファイル。
Tetrads.zip
Object detector用のイネ の四分子画像292個とjsonファイル。