イネ品種のゲノム情報を利用して、
品種改良を加速します!
作物のゲノム研究の最も直接的な応用場面は品種改良です。「10年1品種」「経験と勘」と言われている稲の品種改良のプロセスにゲノム情報を活用することで、変化する地球環境や社会情勢に対応した新品種を迅速につくり出す仕組みを構築できればと考えています。
イネ品種のゲノム情報を利用して、
品種改良を加速します!
作物のゲノム研究の最も直接的な応用場面は品種改良です。「10年1品種」「経験と勘」と言われている稲の品種改良のプロセスにゲノム情報を活用することで、変化する地球環境や社会情勢に対応した新品種を迅速につくり出す仕組みを構築できればと考えています。
- News
岡さん(修士2年生)の発表が第144回講演会日本育種学会優秀発表賞に選出されました
O. sativaとO. glaberrimaの4倍体種間雑種イネで見い出だされた種子および花粉稔性の向上に関わる遺伝領域
☆岡 大晴1, 古田 智敬1, 柏原 壱成1, 牟 竑瑞1, 貴島 祐治2, 長岐 清孝1, 山本 敏央1
(1岡山大・院・環境生命・植物研, 2北海道大・院・農)
関連する育種学会Web site
古田助教の発表が第144回講演会日本育種学会優秀発表賞に選出されました
アジアイネゲノムとの網羅的オーソログ解析に基づくアフリカイネゲノムの特徴づけ
☆古田智敬1, 佐藤 豊2, 吉村 淳3, 芦苅 基行4
(1岡大・資源植物科学研究所, 2遺伝研, 3九大・農学研究院, 4名大・生物機能開発利用研究センター)
関連する育種学会Web site
岡さん(修士2年生)の発表が
第15回中国地域育種談話会
優秀発表賞に選出されました
(2年連続受賞)
イネ種間雑種4倍体の後代分離集団で検出された農業形質関連QTLのdosage効果とヘテロシスの予測
〇岡大晴1・古田智敬1・柏原壱成1・長岐清孝1・貴島祐治2・山本敏央1
(1岡山大学・資源植物科学研究所,2北海道大学)

岡さん(修士1年生)の発表が
第14回中国地域育種談話会
優秀発表賞に選出されました
アジアイネとアフリカイネの稔実4倍体雑種を用いた農業形質に関する遺伝解析法の検討
〇岡大晴1・古田智敬1・柏原壱成1・牟竑瑞1・貴島祐治2・長岐清孝1・山本敏央1
(1岡山大学・資源植物科学研究所,2北海道大学)

古田助教の発表が第141回講演会日本育種学会優秀発表賞に選出されました
他殖性作物の多系交雑集団にも対応した遺伝子型データエラー修正ツール「GBScleanR」の開発
☆古田智敬1,芦苅基行2,山本敏央1
(1岡山大学・資源植物科学研究所,2名古屋大学・生物機能開発利用研究センター)
関連する育種学会Web site
- Research
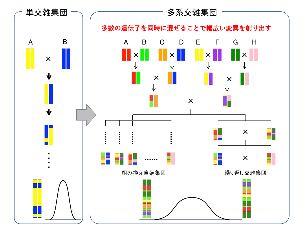
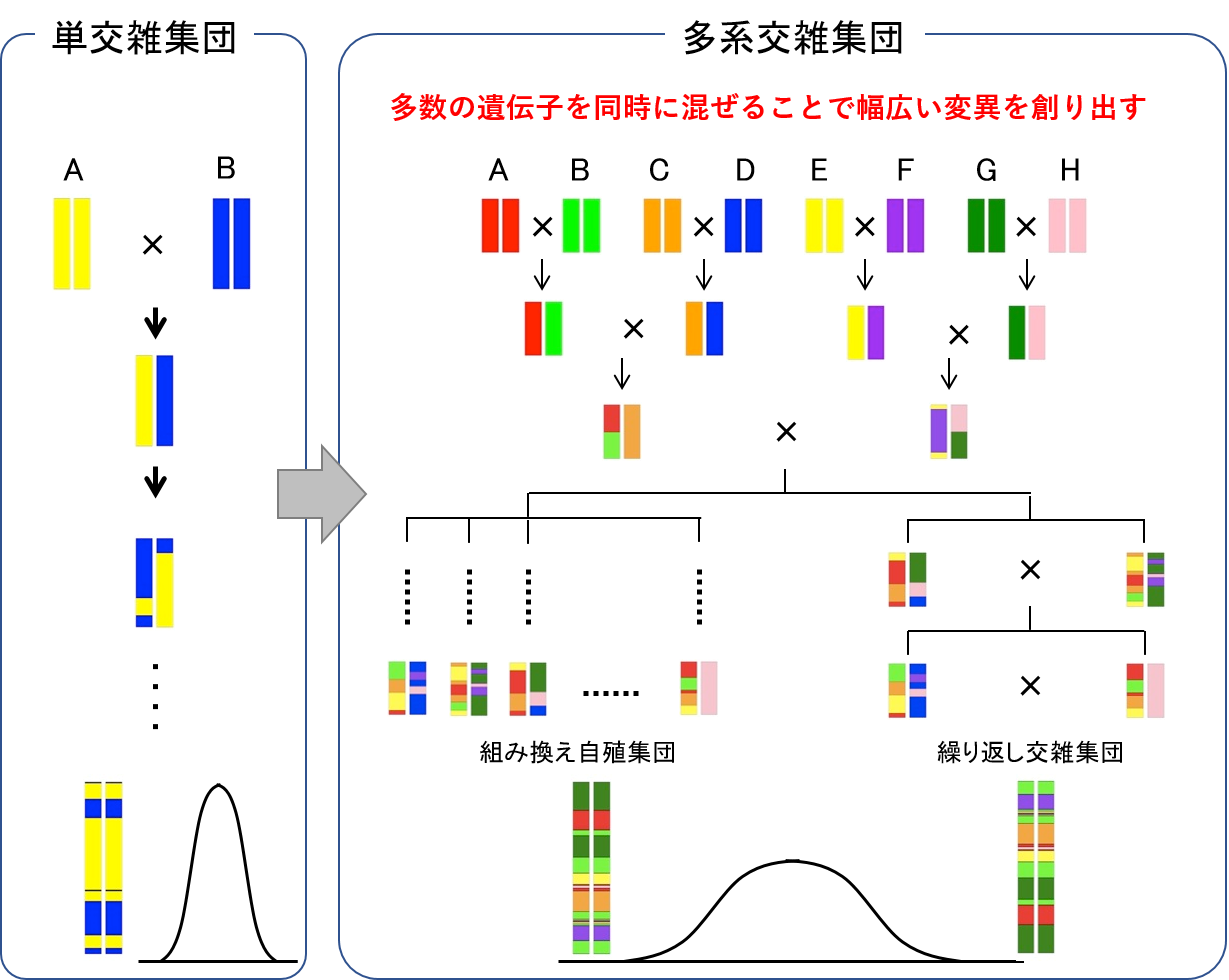
多系交雑に基づく新しいイネ育種法
複数品種の積み上げ交雑からなる多系交雑集団を作出し、環境ストレスに強い遺伝子の探索や、単交雑集団にない新しい変異を生み出す育種集団としての可能性を検証しています。
倍数性を利用した遠縁交雑育種の実現
アジアの栽培イネとアフリカの栽培イネの雑種4倍体系統やその還元2倍体系統のゲノム解析を通じて、不稔や弱勢が障壁となっている遠縁イネ同士の自由な交雑育種の実現を目指しています。

バイオインフォマティクスと統計遺伝学に基づく高効率迅速育種法の開発
大規模な環境変動や急激な人口増加に対応するために、迅速で効率的な作物育種法の開発がますます必要とされています。しかし従来の育種法では、複数回に渡る交配と形質調査を繰り返すため、どうしても時間がかかってしまいます。そこで、これまで蓄積されてきた遺伝学や生理学、作物学などにおける様々なデータとゲノム情報をフル活用し、育種プロセスを短縮する新たな手法の開発を目指しています。

エピジェネティック修飾を見る
植物を形作る個々の細胞は、その役割に応じて、組織内で異なるエピジェネティック修飾パターンをもつと考えられます。しかし、これまでの方法は細胞集団をバルクで解析する為、個々の細胞のエピジェネティック修飾パターンを知ることができませんでした。私たちは植物組織内で細胞の位置情報を維持したまま1細胞レベルでエピジェネティック修飾を解析する方法を開発しています。(画像機械学習については、こちらの特設ページをご覧ください)

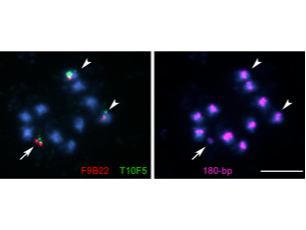
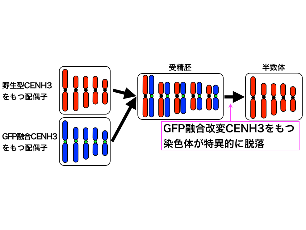
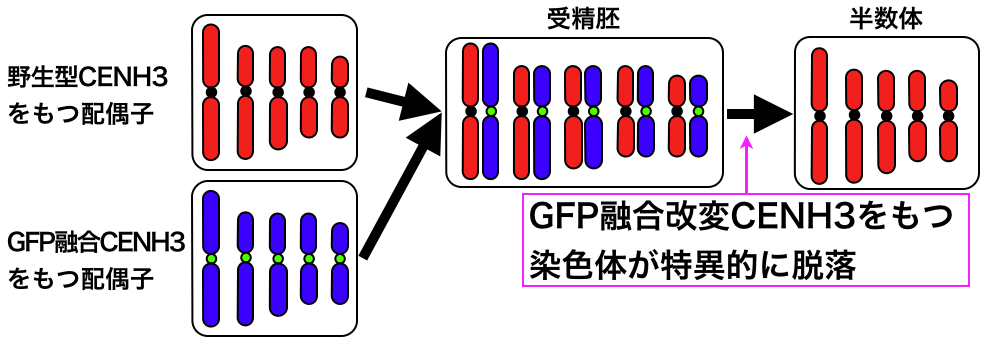
動原体を改変して半数体をつくる
半数体は倍加半数体による育種年限の短期化に利用できますが、半数体が得られる種は限られています。シロイヌナズナやトウモロコシでは、動原体の機能を部分的に失わせた系統と野生型系統を交配すると野生型系統由来の染色体をもつ半数体ができることが報告されました。私たちはこれまでの基礎研究で得られた結果を活かし、これらの種で用いられた方法を簡便化することにより多くの種で利用可能な半数体作出系を開発しています。
植物人工染色体をつくる
染色体の機能要素を含むDNA鎖は、「人工染色体」として機能します。人工染色体には、(1)長いDNAを保持できる、(2)安定な遺伝子発現が保証される、(3)組換えDNAの伝達を制御できる。などの利点があります。最近、私たちのグループでは、トランスポゾンと特異的組換え系を組合わせてシロイヌナズナの環状人工染色体をトップダウン法(通常の染色体を染色体機能要素のみを残して小型化する方法)により作出しました。
- Member

教授:山本 敏央 Prof. YAMAMOTO, Toshio
E-mail:yamamo101040@(@以下はokayama-u.ac.jp を付けてください)
専門分野: 作物育種学
准教授:長岐 清孝 Assoc. Prof. NAGAKI, Kiyotaka
E-mail:nagaki@(@以下はokayama-u.ac.jp を付けてください)
専門分野: 分子細胞遺伝学
助教:古田 智敬 Assist. Prof. FURUTA, Tomoyuki
E-mail:f.tomoyuki@(@以下はokayama-u.ac.jp を付けてください)
専門分野:植物遺伝育種学
技術職員:柏原 壱成 KASHIHARA, Kazunari
- Publications
https://doi.org/10.1093/genetics/iyad055
Kanako Bessho-Uehara, Kengo Masuda, Diane R Wang, Rosalyn B Angeles-Shim, Keisuke Obara, Keisuke Nagai, Riri Murase, Shin-Ichiro Aoki, Tomoyuki Furuta, Kotaro Miura, Jianzhong Wu, Yoshiyuki Yamagata, Hideshi Yasui, Michael B Kantar, Atsushi Yoshimura, Takumi Kamura, Susan R McCouch, Motoyuki Ashikari. Regulator of Awn Elongation 3, an E3 ubiquitin ligase, is responsible for loss of awns during African rice domestication.
Proceedings of the National Academy of Sciences of the United States of America. 120(4).
https://doi.org/10.1073/pnas.2207105120
Asami Michikawa, Moeko Okada, Tatsuya M. Ikeda, Kiyotaka Nagaki, Kentaro Yoshida , Shigeo Takumi. Phenotypic effects of Am genomes in nascent synthetic hexaploids derived from interspecific crosses between durum and wild einkorn wheat. PLoS ONE 18(4): e0284408.
https://doi.org/10.1371/journal.pone.0284408
Potlapalli, B. P., Ishii, T., Nagaki, K., Somasundaram, S., Houben, A. CRISPR-FISH: A CRISPR/Cas9-based labeling method. In Plant Cytogenetics and Cytogenomics. Ed. Heitkam, T., Garcia, S. Humana New York.
https://doi.org/10.1007/978-1-0716-3226-0
Nagata, K., Nonoue, Y., Matsubara, K., Mizobuchi, R., Ono, N., Shibaya, T., Ebana, K., Ogiso-Tanaka, E., Tanabata, T., Sugimoto, K., Taguchi-Shiobara, F., Yonemaru, J., Uga, Y., Fukuda, A., Ueda, T., Yamamoto, S., Yamanouchi, U., Takai, T., Ikka, T., Kondo, K., Hoshino, T., Yamamoto, E., Adachi, S., Sun, J., Kuya, N., Kitomi, Y., Iijima, K., Nagasaki, H., Shomura, A., Mizubayashi, T., Kitazawa, N., Hori, K., Ando, T., Yamamoto, T., Fukuoka, S., Yano, M. Development of 12 sets of chromosome segment substitution lines that enhance allele mining in Asian cultivated rice. Breed. Sci. 73: 332-342.
https://doi.org/10.1270/jsbbs.23006
Kojima, M., Makita, N., Miyata, K., Yoshino, M., Iwase, A., Ohashi, M., Surjana, A., Kudo, T., Takeda-Kamiya, N., Toyooka, K., Miyao, A., Hirochika, H., Ando, T., Shomura, A., Yano, M., Yamamoto, T., Hobo, T., Sakakibara, H. A cell wall-localized cytokinin/purine riboside nucleosidase is involved in apoplastic cytokinin metabolism in Oryza sativa. Proc. Natl. Acad. Sci. U S A. 120: e2217708120.
https://doi.org/10.1073/pnas.2217708120
https://thericejournal.springeropen.com/articles/10.1186/s12284-022-00560-w
Masahiro Yamashita, Chikako Ootsuka, Hikaru Kubota, Shunsuke Adachi, Takuya Yamaguchi, Kazumasa Murata, Toshio Yamamoto, Tadamasa Ueda, Taiichiro Ookawa, Tadashi Hirasawa. Alleles of high-yielding indica rice that improve root hydraulic conductance also increase flag leaf photosynthesis, biomass, and grain production of japonica rice in the paddy field. Field Crops Research 289: 108725
https://www.sciencedirect.com/science/article/abs/pii/S0378429022002969
石井孝佳・長岐清孝・菊池真司 細胞遺伝学の新潮流-より速く、より広く、より細かく、そして創出へ、古くて新しいゲノムの見える化技術. 化学と生物 58: 606-613.
山本敏央・古田智敬・小川大輔・米丸淳一・國吉大地・貴島祐治 イネ育種における遺伝的多様性を拡大するための2,3のアプローチ. 作物研究 65:83-87.
Inoue, K., Takahagi, K., Kouzai, Y., Koda, S., Shimizu, M., Uehara-Yamaguchi, Y., Nakayama, R., Kita, T., Onda, Y., Nomura, T., Matsui, H., Nagaki, K., Nishii, R. and Mochida, K. Parental legacy and regulatory novelty in Brachypodium diurnal transcriptomes accompanying their polyploidy. NAR Genomics and Bioinformatics 2: 1-14.
Nagai, K., Mori, Y., Ishikawa, S., Furuta, T., Gamuyao, R., Niimi, Y., Hobo, T., Fukuda, M., Kojima, M., Takebayashi, Y., Fukushima, A., Himuro, Y., Kobayashi, M., Ackley, W., Hisano, H., Sato, K., Yoshida, A., Wu, J., Sakakibara, H., Sato, Y., Tsuji, H., Akagi, T., Ashikari, M. Antagonistic regulation of the gibberellic acid response during stem growth in rice. Nature 584 :109-114. doi: 10.1038/s41586-020-2501-8.
Nagaki, K. and Yamaji, N. Decrosslinking enables visualization of RNA-guided endonuclease–in situ labeling signals for DNA sequences in plant tissues. Journal of Experimental Botany 71(6): 1792-1800
San, N. S., Suzuki, K., Soda, K., Adachi, S., Kasahara, H., Yamamoto, T., Ikka, T., Kondo, K., Yamanouchi, U., Sugimoto, K., Nagamura, Y., Hirasawa, T. and Ookawa, T. Semi-dwarf 1 (sd1) gene enhances light penetration into the canopy through regulating leaf inclination angle in rice. Field Crops Research 246: 107694
Ogawa, D., Sakamoto, T., Tsunematsu, H., Yamamoto, T., Kanno, N., Nonoue, Y. and Yonemaru, J. Surveillance of panicle positions by unmanned aerial vehicle to reveal morphological features of rice. PLoS ONE 14(10): e0224386
Nomura, T., Arakawa, N., Yamamoto, T., Ueda, T., Adachi, S., Yonemaru, J., Abe, A., Takagi, H. and Ookawa, T. Next generation long-culm rice with superior lodging resistance and high grain yield, Monster Rice 1. PLoS ONE 14(8): e0221424
Adachi, S., Yamamoto, T., Nakae, T., Yamashita, M., Uchida, M., Karimata, R., Ichihara, N., Soda, K., Ochiai, T., Ao, R., Otsuka, C., Nakano, R., Takai, T., Ikka, T., Kondo, K., Ueda, T., Ookawa, T. and Hirasawa, T. Genetic architecture of leaf photosynthesis in rice revealed by different types of reciprocal mapping populations. Journal of Experimental Botany 70(19): 5131-5144
Sun, J., Ma, D., Tang, L., Zhao, M., Zhang, G., Wang, W., Song, J., Li, X., Liu, Z., Zhang, W., Xu, Q., Zhou, Y., Wu, J., Yamamoto, T., Dai, F., Li, S., Zhou, G., Zheng, H., Xu, Z. and Chen, W. Population Genomic Analysis and De novo Assembly Reveal the Origin of Weedy Rice as an Evolutionary Game. Mol. Plant 12(5): 632-647
Samadi, A. F., Suzuki, H., Ueda, T., Yamamoto, T., Adachi, S. and Ookawa, T. Identification of quantitative trait loci for breaking and bending types lodging resistance in rice, using recombinant inbred lines derived from Koshihikari and a strong culm variety, Leaf Star. Plant Growth Regulation 89(1): 83-98
Ogawa, D., Yamamoto, E., Ootani, T., Kanno, N., Tsunematsu, H., Nonoue, Y., Yano, M., Yamamoto. T. (co-corresponding), and Yonemaru, J. 2018. Haplotype-based allele mining with JAPAN MAGIC population in rice. Scientific reports, 8: 4379
San, N.S., Ootsuki, Y., Adachi, S., Yamamoto, T., Ueda, T., Tanabata, T., Motobayashi, T., Ookawa, T., and Hirasawa, T. 2018. A near-isogenic rice line carrying a QTL for larger leaf inclination angle yields heavier biomass and grain. Field Crops Research, 219:131-138
Kobayashi, A., Hori, K., Yamamoto, T. (corresponding), and Yano, M. 2018. Koshihikari: a premium short-grain rice cultivar – its expansion and breeding in Japan. Rice 11: 15
Reuscher, S., Furuta, T., Bessho-Uehara, K., Cosi, M., Jena, K.K., Toyoda, A., Fujiyama, A., Kurata, N., Ashikari, M. 2018. Assembling the genome of the African wild rice Oryza longistaminata by exploiting synteny in closely related Oryza species. Communications biology, 1: 162
Kuroha, T., Nagai, K., Gamuyao, R., Wang, D.R., Furuta, T., Nakamori, M., Kitaoka, T., Adachi, K., Minami, A., Mori, Y., Mashiguchi, K., Seto, Y., Yamaguchi, S., Kojima, M., Sakakibara, H., Wu, J., Ebana, K., Mitsuda, N., Ohme-Takagi, M., Yanagisawa, S., Yamasaki, M., Yokoyama, R., Nishitani, K., Mochizuki, T., Tamiya, G., McCouch, S.R., Ashikari, M. 2018. Ethylene-gibberellin signaling underlies adaptation of rice to periodic flooding. Science, 361(6398): 181-186
Nakano, H., Yoshinaga, S., Takai, T., Arai-Sanoh, Y., Kondo, K., Yamamoto, T., Sakai, H., Tokida, T., Usui, Y., Nakamura, H., Hasegawa, T., and Kondo, M. 2017. Quantitative trait loci for large sink capacity enhance rice grain yield under free-air CO2 enrichment conditions. Scientific reports 7: 1827
Yamamoto, T., Suzuki, T., Suzuki, K., Adachi, S., Sun, J., Yano, M., Ookawa, T., and Hirasawa, T. 2017. Characterization of a genomic region that maintains chlorophyll and nitrogen contents during ripening in a high-yielding stay-green rice cultivar. Field Crops Research 206: 54-64
Adachi, S., Yoshikawa, K., Yamanouchi, U., Tanabata, T., Sun, J., Ookawa, T., Yamamoto, T., Sage, R.F., Hirasawa, T., and Yonemaru, J. 2017. Fine mapping of Carbon Assimilation Rate 8, a quantitative trait locus for flag leaf nitrogen content, stomatal conductance and photosynthesis in rice. Frontiers in plant science 8: 60
Bessho-Uehara, K., Furuta, T., Masuda, K., Yamada, S., Angeles-Shim, R.B., Ashikari, M., Takashi, T. 2017. Construction of rice chromosome segment substitution lines harboring Oryza barthii genome and evaluation of yield-related traits.
Furuta, T., Ashikari, M., Jena, K.K., Doi, K., Reuscher, S. 2017. Adapting genotyping-by-sequencing for rice F2 populations. G3, 7(3): 881-893
Yamamoto, T., Suzuki, T., Suzuki, K., Adachi, S., Sun, J., Yano, M., Ookawa, T., and Hirasawa, T. 2016. Detection of QTL for exudation rate at ripening stage in rice and its contribution to hydraulic conductance. Plant Science 242: 270-277
山本敏央(2016)バイオマス増大に向けたイネ次世代育種法の開発と利用 JATAFFジャーナル Vol.4(5):65
Furuta, T., Uehara, K., Angeles-Shim, R.B., Shim, J., Nagai, K., Ashikari, M., Takashi, T. 2016. Development of chromosome segment substitution lines harboring Oryza nivara genomic segments in Koshihikari and evaluation of yield-related traits. Breeding science, 66(5): 845-850
Bessho-Uehara, K., Wang, D.R., Furuta, T., Minami, A., Nagai, K., Gamuyao, R., Asano, K., Angeles-Shim, R.B., Shimizu, Y., Ayano, M., Komeda, N., Doi, K., Miura, K., Toda, Y., Kinoshita, T., Okuda, S., Higashiyama, T., Nomoto, M., Tada, Y., Shinohara, H., Matsubayashi, Y., Greenberg, A., Wu, J., Yasui, H.,Yoshimura, A., Mori, H., McCouch, S.R., Ashikari, M. 2016. Loss of function at RAE2, a previously unidentified EPFL, is required for awnlessness in cultivated Asian rice. PNAS, 113(32): 8969-8974
Ramos, J.M., Furuta, T., Uehara, K., Chihiro, N., Angeles-Shim, R.B., Shim, J., Brar, D.S., Ashikari, M., Jena, K.K. 2016. Development of chromosome segment substitution lines (CSSLs) of Oryza longistaminata A. Chev. & Röhr in the background of the elite japonica rice cultivar, Taichung 65 and their evaluation for yield traits. Euphytica, 210(2): 151-163
Kurokawa, Y., Noda, T., Yamagata, Y., Angeles-Shim, R., Sunohara, H., Uehara, K., Furuta, T., Nagai, K., Jena, K.K., Yasui, H., Yoshimura, A., Ashikari, M., Doi, K. 2016. Construction of a versatile SNP array for pyramiding useful genes of rice. Plant science, 242: 131-139
Wada, T., Miyahara, K., Sonoda, J., Tsukaguchi, T., Miyazaki, M., Tsubone, M., Ando, T., Ebana, K., Yamamoto, T., Iwasawa, N., Umemoto, T., Kondo, M., and Yano, M. 2015. Detection of QTLs for white-back and basal-white grains caused by high temperature during ripening period in japonica rice. Breeding Science 65(3): 216-225
Yano, K., Ookawa, T., Aya, K., Ochiai, Y., Hirasawa, T., Ebitani, T., Takarada, T., Yano, M., Yamamoto, T., Fukuoka, S., Wu, J., Ando, T., Ordonio, R.L., Hirano, K., and Matsuoka, M. 2015. Isolation of a novel lodging resistance QTL gene involved in strigolactone signaling and its pyramiding with a QTL gene involved in another mechanism. Molecular Plant 8(2): 303-314
Habu, Y., Ando, T., Ito, S., Nagaki, K., Kishimoto, N., Taguchi-Shiobara, F., Numa, H., Yamaguchi, K., Shigenobu, S., Murata, M., Meshi, T., and Yano, M. 2015. Epigenomic modification in rice controls meiotic recombination and segregation distortion. Molecular Breeding 35: 644-653
Furuta, T., Komeda, N., Asano, K., Uehara, K., Gamuyao, R., Angeles-Shim, R.B., Nagai, K., Doi, K., Wang, D.R., Yasui, H., Yoshimura, A., Wu, J., McCouch, S.R., Ashikari, M. 2015. Convergent loss of awn in two cultivated rice species Oryza sativa and Oryza glaberrima is caused by mutations in different loci. G3, 5(11): 2267-2274
Song, X.J., Kuroha, T., Ayano, M., Furuta, T., Nagai, K., Komeda, N., Segami, S., Miura, K., Ogawa, D., Kamura, T., Suzuki, T., Higashiyama, T., Yamasaki, M., Mori, H., Inukai, Y., Wu, J., Kitano, H., Sakakibara, H., Jacobsen, S.E., Ashikari, M. 2015. Rare allele of a previously unidentified histone H4 acetyltransferase enhances grain weight, yield, and plant biomass in rice. PNAS, 112(1): 76-81
Tek, A. L., Kashihara, K., Murata, M., Nagaki, K. 2014. Identification of the centromere-specific histone H3 variant in Lotus japonicus. Gene 538: 8-11
山本敏央 (2014) 多収稲の光合成能力に貢献するGPS遺伝子を発見 ニューカントリー 61(8):50-51
山本敏央, 髙井俊之 (2014) 多収イネの光合成能力に貢献する遺伝子の特定 明日の食品産業 (448):35-39
Furuta, T., Uehara, K., Angeles-Shim, R.B., Shim, J., Ashikari, M., Takashi, T. 2014. Development and evaluation of chromosome segment substitution lines (CSSLs) carrying chromosome segments derived from Oryza rufipogon in the genetic background of Oryza sativa L. Breeding science, 63(5): 468-475
Shibata, F., Nagaki, K., Yokota, E., Murata, M. 2013. Tobacco karyotyping by accurate centromere identification and novel repetitive DNA localization. Chromosome Res. 21: 375-381
安部匡, 倉俣正人, 岩崎行玄, 本間利光, 茨木俊行, 山本敏央, 矢野昌裕, 村上政治, 石川覚 (2013) ガンマ線照射による突然変異育種法を用いた難脱粒性カドミウムファイトレメディエーション用イネ系統「MJ3」および「MA22」の育成 育種学研究 15(2): 17-24
2011年以前の論文については、コチラをご覧ください。
山本敏央教授の全論文リスト(Research mapへのリンク)
長岐清孝准教授の全論文リスト(Elsevier Pureへのリンク)
古田智敬助教の全論文リスト(Research mapへのリンク)
- 生物学教材
・はじめての染色体〜染色体写真集&実験マニュアル〜
PLOS ONE(2012年12月7日号)で発表した研究結果(タマネギの細胞分裂の3D詳細解析)を中心とした私たちの成果に基づいた電子書籍です。最新の3D解析データー画像に加えてインタラクティブな3Dモデルなども多数掲載しており、生物の副教材としてもご利用いただけるかと思います。この教材には、iPadおよびOSX用のiBooks版、インターネット用のブラウザーでも閲覧可能なWeb版、および動画は動かないけれどオフラインで使えて汎用性もあるPDF版があります。
iPad用のiBooks版では、インタラクティブに学習することができます。できるだけ鮮明な画像をお届けするために高解像度画像を使用していますので、iPadのバージョンによっては読み込みに時間がかかるる場合があります。こちらのテストではiPad2以降の機種(iPad2、第3世代、第4世代、iPad mini)では快適に利用できました。また、初代iPadでも3Dモデルの表示に3秒程度かかる場合もありましたが、作動はしました。残念ながら、Appleの仕様で「インタラクティブなiBooksファイル」は、iPhoneおよびiPod touchでは作動しません。
Web版は、iBooks版をWebで試用できるようにダウンコンバートしたものであり、iBooks版と比較して機能が縮小されている部分があります(インタラクティブなことができません)。PDFは印刷用のフォーマットなので、PDF版では動画や3Dモデルは動きません。
*「指導者用のデジタル教材」等としてコンピューターを用いてフルサイズの動画を表示する必要がある方は、PLOS ONEの論文のFigure S3(間期)、Figure S4(前期)、Figure S6(中期)、Figure S7(後期)、Figure S8(終期)、Figure S9(後終期)をご利用ください。「Supporting Information」の部分に上記の動画へのリンクがあります。PLOS ONEは、どなたでも無料で閲覧可能です。
はじめての染色体 for iPad & OSX iBooks (おためし版)
iPadで直接ダウンロードするか、コンピューターでダウンロードした後にiTunesを使ってiPadに転送してください。iOS App iBooksを使用して読むことができます。
HajimeteOtameshi.ibooks←はじめての染色体(おためし版)をダウンロード(20.3 MB)
はじめての染色体(PDF版)
PDFフォーマットですので多くの電子書籍リーダーやコンピューター、タブレット、スマートフォンでiPadに表示されるものに近い形で御覧になれます。また、Web版とは異なり、オフラインでも利用できます。ただし、「連続写真」や「3Dモデル」は動きません。連続写真の一部については、上記「指導者用のデジタル教材」のところで説明してある、PLOS ONEの論文のFigure S3&4、S6〜9の動画をあらかじめダウンロードしておけば、iPad以外の端末でもiPad版に近い品質で利用することができます。
HajimeteJAB4.pdf←はじめての染色体(PDF版)をダウンロード(31.1 MB)
はじめての染色体 nano(PDF版、折りたたみハンドブック)
両面印刷して折りたたむとハンドブックができあがります。軽い気持ちで作りましたが、けっこう便利かも。
Hajimetenano.pdf←はじめての染色体 nano をダウンロード(36 MB)