
研究組織
光環境適応研究グループ
 |
光環境適応研究グループのホームページへ |
教員
 |
教授: 坂本 亘 Prof. Dr. Wataru SAKAMOTO E-mail:saka@(@以下はokayama-u.ac.jp を付けてください。) 専門分野: 植物分子生物学・遺伝学 |
||
 |
准教授: 松島 良 Assoc. Prof. Dr. Ryo MATSUSHIMA E-mail:rmatsu@(@以下はokayama-u.ac.jp を付けてください。) 専門分野: 細胞生物学 |
||
 |
准教授: 桶川 友季 Assoc. Prof. Dr. Yuki OKEGAWA E-mail:okegawa@(@以下はokayama-u.ac.jp を付けてください。) 専門分野: 植物生理学 |
主な研究テーマ
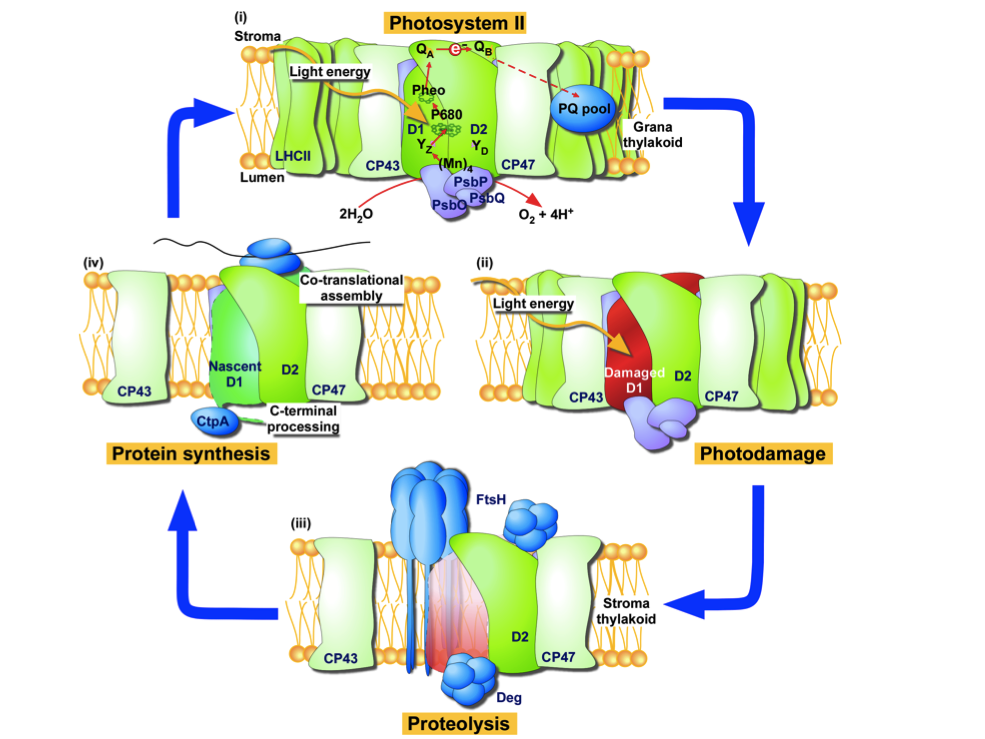
| 1. 強光ストレス下における植物の光障害適応機構の解析 光合成において過剰な光エネルギーは光化学系IIに障害をあたえ、光合成機能の低下を引き起こすことが知られている。これを回避するため、光化学系IIでは障害をうけた反応中心タンパク質D1を直ちに分解/修復し、系全体の機能維持を行っている。光化学系IIの迅速な修復に関わる因子について、プロテアーゼを中心に解析している。 |
 |
| 2. 葉緑体膜の修復と維持に関わる新奇タンパク質の解析 葉緑体は過剰な光エネルギーで脂質やタンパク質が損傷を受けやすいため、それらを緩和して環境に適応するための様々なしくみを発達させている。損傷を受けた葉緑体の膜を修復しながら葉緑体機能維持に関わっている、VIPP1と呼ばれるタンパク質について解析している。このタンパク質を強化することで、葉緑体での光合成能を強化させ、環境ストレスに強い作物の育成を目指している。 |
 |
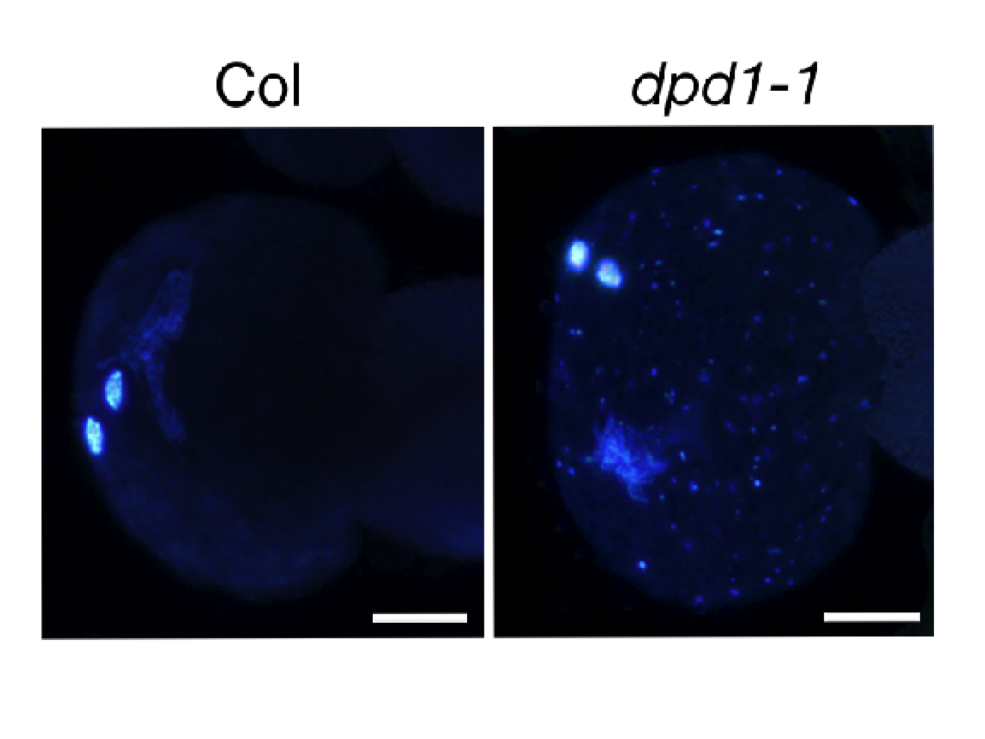
| 3. 葉緑体DNAの代謝機構に関する研究 細胞内共生に由来して葉緑体が持つDNAは、植物の発生段階によってそのコピー数が変動するがその生理学的意義や分子機構については不明である。これらの疑問を明らかにするための研究を、シロイヌナズナ突然変異体を用いて行っており、オルガネラDNA分解酵素の同定によるDNA分解現象の発見と、その機能解析を行っている。 |
 |
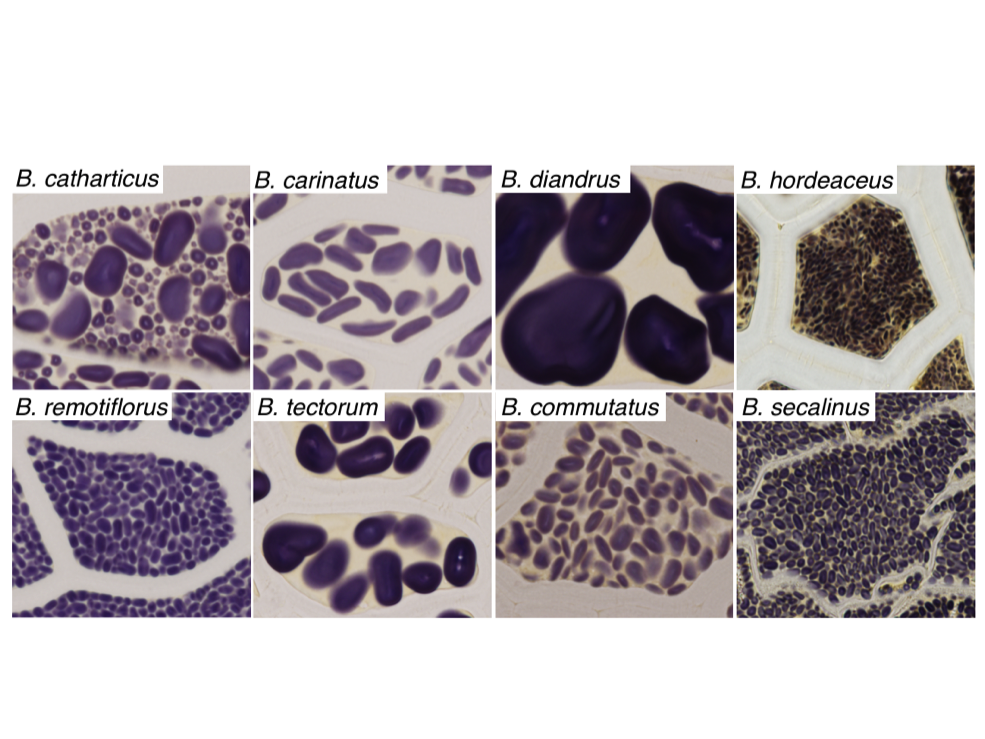
| 4. 澱粉粒の形状多様性を支配する分子機構の解析 澱粉粒は、植物が光合成産物として色素体内に蓄積するグルコース多量体である。澱粉粒の形状は植物種によって大きく異なるが、その形状多様性を支配する分子機構は現在まで不明である。澱粉粒の形状について、イネ突然変異体を単離し解析を行っている。 |
 |
Latest publications (for complete and most current publications visit group pages)
(1) Ozawa S.I, Zhang G, Sakamoto W. Dysfunction of Chloroplast Protease Activity Mitigates pgr5 Phenotype in the GreenAlgae Chlamydomonas reinhardtii. Plants (Basel) 13: 606. doi.org/10.3390/plants13050606 (2024. 2.)
(2) 小川 由 シロイヌナズナのFZL タンパク質による葉緑体膜リモデリングの遺伝生理学的解析.博士学位論文 (岡山大学) (2024. 3.)
(3) Sakamoto W, Takami T. Plastid Inheritance Revisited: Emerging Role of Organelle DNA Degradation in Angiosperms. Plant Cell Physiol. 65: 484-492. doi.org/10.1093/pcp/pcad104 (2024. 4.)
(4) Kumari P, Matsushima R, Hirayama T, Mikami K. Responses of the marine filamentous red alga ʻBangiaʼ sp. ESS1 to recurrent changes in seawater concentration. Algal Res. 80: 103551. doi.org/10.1016/j.algal.2024.103551 (2024. 5.)
(5) Islam M.F, Yamatani H, Takami T, Kusaba M, Sakamoto W. Characterization of organelle DNA degradation mediated by DPD1 exonuclease in the rice genome-edited line. Plant Mol. Biol. 114: 71. doi.org/10.1007/s11103-024-01452-x (2024. 6.)
(6) Matsushima R, Hisano H, Kim J.S, McNelly R, Oitome N.F, Seung D, Fujita N, Sato, K. Mutations in starch BRANCHING ENZYME 2a suppress the traits caused by the loss of ISOAMYLASE1 in barley. Theor. Appl. Genet. 137: 212. doi.org/10.1007/s00122-024-04725-7 (2024. 8.)
(7) 加藤裕介・坂本 亘 光合成反応における光損傷と修復のメカニズムの解明. 光アライアンス 35: 9-12. (2024.9.)
(8) Islam M.F. Studies on the role of organelle DNA degradation mediated by DPD1 nuclease in rice. 博士学位論文 (岡山大学)(2024. 9.)
(9) Brunje A, Fussl M, Eirich J, Boyer J, Heinkow P, Neumann U, Konert M, Ivanauskaite A, Seidel J, Ozawa S, Sakamoto W, Meinnel T, Schwarzer D, Mulo P, Giglione C, Finkemeier I. The plastidial protein acetyltransferase GNAT1 forms a complex with GNAT2, yet their interaction is dispensablefor state transitions. Mol. Cell Proteomics. 23: 100850. doi.org/10.1016/j.mcpro.2024.100850 (2024. 11.)





