
研究組織
環境応答機構研究グループ
 |
環境応答機構研究グループのホームページへ |
教員
 |
教授: 平山 隆志 Prof. Dr. Takashi HIRAYAMA E-mail: hira-t@(@以下はokayama-u.ac.jp を付けてください。) 専門分野: 植物分子遺伝学、植物分子生物学 |
||
 |
准教授: 森 泉 Assoc. Prof. Dr. Izumi MORI E-mail:imori@(@以下はokayama-u.ac.jp を付けてください。) 専門分野: 植物生理学 |
||
 |
准教授: 池田 陽子 Assoc. Prof. Dr. Yoko IKEDA E-mail:yikeda@(@以下はokayama-u.ac.jp を付けてください。) 専門分野:植物分子遺伝学 |
主な研究テーマ
| 1. 高等植物の環境応答機構の解明 高等植物の環境認識機構、環境情報の統合機構、更に環境の変化に対する応答の統御機構を理解したいと考えている。そのため、主にモデル植物を用いた分子遺伝学的、分子生物学的解析を行なっている。特に非生物的ストレス応答に関与する植物ホルモンのアブシジン酸に対する応答の分子機構の解明と、アブシジン酸応答に様々な形で関わる細胞、組織、そして個体レベルの生理機能について研究を進めている。 |
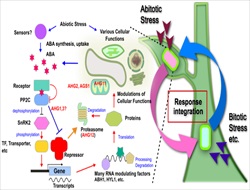 |
| 2. 気孔の環境応答機構の解明 気孔は光に応答して開き,乾燥や菌類の侵入刺激に応答して閉じるなどの運動をする.この過程において,孔辺細胞内で環境情報が気孔運動に変換される機構の解明に取り組んでいる. |
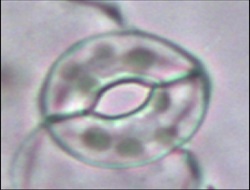 |
| 3. 環境に応じたエピジェネティックな遺伝子発現調節機構の研究 生命の遺伝情報をつかさどる遺伝子は、DNA塩基配列にコードされているが、遺伝子が発現し、機能するためには、DNAメチル化やヒストン修飾といったDNA塩基配列以外のエピジェネティックな情報が重要な役割を果たしていることが明らかになっている。これらの調節機構が成長段階や環境変化に応じて働く機構について研究を行っている。 |
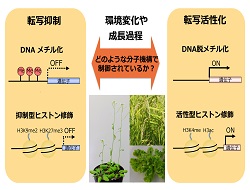 |
| 4. 穂発芽抵抗性白粒コムギの解析 コムギは、種子色により赤粒と白粒があり、白粒コムギは穂発芽しやすく国内では収穫期が梅雨と重なり穂発芽が多発するため品質が悪い。様々な交配組み合わせより強い種子休眠能力を有する穂発芽抵抗性白粒コムギ1-117系統を見いだした。分子遺伝学的解析や植物ホルモンの測定等を行い1-117系統の持つ強い種子休眠能力を明らかにしようと研究を行っている。 |
 |
Latest publications (for complete and most current publications visit group pages)
(1) Himi E, Kurihara-Yonemoto S, Abe F, Takahashi H, Tanaka K, Matsuura T, Maekawa M, Sasaki T, Rikiishi K. Tamyb10‑D1 restores red grain color and increases grain dormancy via suppressing expression of TaLTP2.128, non‑specific lipid transfer protein in wheat. Euphytica. 220: 16. doi.org/10.1007/s10681-023-03265-3 (2024. 1.)
(2) Kumari P, Matushima R, Hirayama T, Mikami K. Responses of the marine filamentous red alga ʻBangiaʼ sp. ESS1 to recurrent changes in seawater concentration, Algal Research 80: 103551. doi.org/10.1016/j.algal.2024.10355 (2024. 6.)
(3) Unung O.O, Bensedira H.E.S, Matsuura T, Mori I.C, Shimomura Y, Yaeno T, Kaya H, Kobayashi, K. Possible roles of immunity-related response in modulating chlorosis induced by the silencing of chloroplast HSP90C in tobacco models. J. Gen. Plant Pathol. 90: 298-308. doi.org/10.1007/s10327-024-01191-3 (2024. 7.)
(4) 平山隆志 成長過程の理解と農業形質予測.アグリバイオ 8: 34-38. (2024. 7.)
(5) 池田陽子・植村美代子・森 泉 第4章 ChIP 用調整プロコトール 2植物組織② ChIP-seq とその応用編(カキ).誰でも再現できるNGS「前」サンプル調整プロトコール 実験医学別冊 ISBN 978-4-7581-2272-6 (2024. 7.)
(6) Hsiang T.F, Chen Y.Y, Nakano R, Oikawa A, Matsuura T, Ikeda Y, Yamane H. Dormancy regulator Prunus mume DAM6 promotes ethylene-mediated leaf senescence and abscission. Plant Mol. Biol. 114: 99. doi.org/10.1007/s11103-024-01497-y (2024. 9.)
(7) Mozhgani M, Ooi L, Espagne C, Filleur S, Mori I.C. Cytosolic acidification and oxidation are the toxic mechanisms of SO2 in Arabidopsis guard cells. Biosci. Biotechnol. Biochem. 88: 1164-1171. doi.org/10.1093/bbb/zbae092 (2024. 10.)
(8) Grossi C.E.M, Tani A, Mori I.C, Matsuura T, Ulloa R.M. Plant Growth-Promoting Abilities of Methylobacterium sp. 2A Involve Auxin-Mediated Regulation of the Root Architecture Plant Cell Environ. 47: 5343-5357. doi.org/10.1111/pce.15116 (2024. 12.)
(9) Mori A, Nakagawa S, Suzuki T, Suzuki T, Gaudin V, Matsuura T, Ikeda Y, Tamura K. The importin α proteins IMPA1, IMPA2, and IMPA4 play redundant roles in suppressing autoimmunity in Arabidopsis thaliana. Plant J. doi.org/10.1111/tpj.17203 (2024. 12. Online preview)
(10) Nakamura K, Kikuchi Y, Shiraga M, Kotake T, Hyodo K, Taketa S, Ikeda Y. SHORT AND CROOKED AWN, Encoding an Epigenetic Regulator EMF1, Promotes Barley Awn Development. Plant Cell Physiol. doi.org/10.1093/pcp/pcae150 (2024. 12. Online preview)





