
研究組織
ゲノム多様性グループ
| ゲノム多様性グループのホームページへ |
教員
 |
教授: 久野 裕 Prof. Dr. Hiroshi HISANO E-mail: hiroshi.hisano@(@以下はokayama-u.ac.jp を付けてください。) 専門分野: 植物分子育種、植物遺伝資源 |
||
 |
准教授: 最相 大輔 Assoc. Prof. Dr. Daisuke SAISHO E-mail: saisho@(@以下はokayama-u.ac.jp を付けてください。) 専門分野: 植物育種、植物分子遺伝、集団遺伝 |
||
 |
助教: 山森 晃一 Assist Prof. Dr. Koichi YAMAMORI E-mail: k.yamamori@(@以下はokayama-u.ac.jp を付けてください。) 専門分野: 植物育種 |
主な研究テーマ
| 1. オオムギ遺伝資源の評価と利用 当研究所では、世界中で収集されたオオムギとその近縁種の種子を保存しています。私たちの研究グループは、それらのオオムギ遺伝資源を用いて、農業形質の評価やゲノム配列解読による多様性解析を行っています。それらの研究を通して、土壌ストレス適応性、病害抵抗性などの農業上重要な遺伝子の単離と機能解析を行うことにより、遺伝資源の産業利用を図っています。 |
 |
| 2. オオムギのゲノム情報解析 私たちのグループでは、植物ストレス科学や育種に役立てることを目的として、オオムギ品種・系統のゲノム配列あるいは発現遺伝子配列の取得と解析を行っています。特にゲノム全体の遺伝子多型情報を活用して、目的の形質をDNA鑑定によって選抜する技術の開発を目指しています。これらの研究を進めるための高速解析ツールやデータベースの開発も行っています。 |
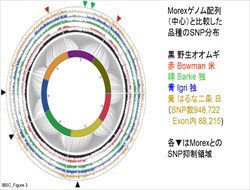 |
| 3. オオムギの遺伝的多様性と適応分化に関する研究 オオムギは中東で起源し、各地の気候や土壌でも生育できるよう分化して、世界中に栽培域を広げました。私たちのグループでは、栽培オオムギの適応分化の遺伝的背景を明らかにすることを目的として、オオムギ遺伝資源の多様性解析研究に取り組んでいます。特に、出穂特性や種子休眠性などの地域適応に関する表現型変異の全体像と、遺伝的分化との関係に注目して研究しています。 |
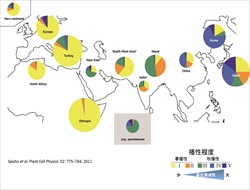 |
| 4. オオムギの有用形質遺伝子の改変と育種への応用 私たちの研究グループでは、遺伝子機能の迅速解析を目的として、オオムギの遺伝子改変技術の高度化を進めています。オオムギの形質転換効率に関わる培養特性関連遺伝子の解析や遺伝解析を行っています。CRISPR/Cas9法などによる標的変異導入(ゲノム編集)技術を効率化し、多様な遺伝子のノックアウトラインの作成を行っています。 |
 |
Latest publications (for complete and most current publications visit group pages)
(1) Hisano H, Hoffie R.E, Kumlehn J, Sato K. Targeted Modification of Grain Dormancy Genes in Barley. In Seed Dormancy: Methods and Protocols, Ed. by N. Kawakami and K. Sato. Springer Nature pp. 149-161. doi.org/10.1007/978-1-0716-3965-8_14 (2024.7.)
(2) Abe F, Kamiya Y, Ishida Y, Hisano H, Kawaura K, Komari T, Sato K. Genome Editing to Produce Knockout Mutations of Seed Dormancy Genes in Wheat. In Seed Dormancy: Methods and Protocols, Ed. by N. Kawakami and K. Sato. Springer Nature pp. 137-148. doi.org/10.1007/978-1-0716-3965-8_13 (2024. 7.)
(3) Matsushima R, Hisano H, Kim J.S. McNelly R, Oitome N.F, Seung D, Fujita N, Sato K. Mutations in starch BRANCHING ENZYME 2a suppress the traits caused by the loss of ISOAMYLASE1 in barley. Theor. Appl. Genet. 137: 212. doi.org/10.1007/s00122-024-04725-7 (2024. 8.)
(4) Kishi-Kaboshi M, Abe F, Chono M, Yamaji N, Sato, K. Evaluation of grain dormancy under field conditions in a wheat (Triticum aestivum) qsd1 triple mutant. Crop Science 2024: 1-11. doi.org/10.1002/csc2.21403 (2024. 11.)





